Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
Mrs.who
Nuovo Arrivato

13 Messaggi |
 Inserito il - 23 maggio 2017 : 14:31:23 Inserito il - 23 maggio 2017 : 14:31:23



|
Buongiorno a tutti, vorrei gentilmente chiedervi un aiuto!
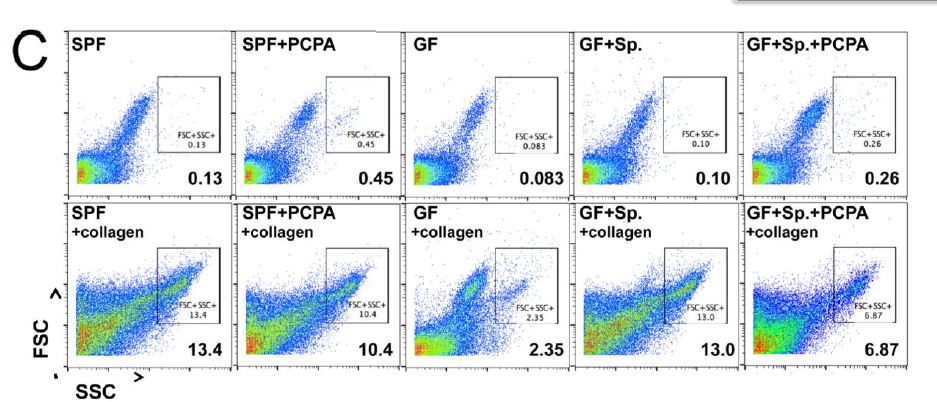
In un articolo scientifico che sto studiando viene valutata la attivazione e aggregazione piastrinica mediante citometria a flusso, e come risultato si ottiene quanto ho allegato:
Immagine:
93,05 KB
didascalia: "Representative flow cytometry plots of large, high granularity (FSChigh, SSChigh) activated platelets after collagen stimulation (bottom), as compared to unstimulated controls (top)."
Qualcuno mi spiega come si interpreta? Ho cercato ovunque ma nessuna spiegazione riesce a farmi comprendere come si interpreta
grazie a tutti!
|
|
|
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
 Inserito il - 23 maggio 2017 : 19:00:22 Inserito il - 23 maggio 2017 : 19:00:22




|
Ciao.
Premetto che non conosco la biologia delle piastrine a sufficienza per essere di grande aiuto ma posso dirti cosa significano i grafici.
Do per scontato che tu sappia a grandi linee come funziona un citofluorimetro. In questo caso devi guardare i parametri in ascissa (SSC) e oridnata (FSC). SSC sta per side scatter e rappresenta la complessità strutturale (più o meno) dell'evento letto dal citofluorimetro, FSC sta per front scatter e rappresenza la dimensione dell'evento.
I grafici che mostri rappresentano, quindi, l'aspetto delle piastrine prima (in alto) e dopo (in basso) attivazione con collagene, in diverse condizioni (SPF, SPF+PCPA, etc., che non ho idea di cosa siano).
Il riquadro nei grafici rappresenta la percentuale di piastrine che sono attivate nelle diverse condizioni. Senza altre informazioni sull'esperimento non posso dirti il significato di tutto ciò.
Spero di essere stato d'aiuto. Chiedi se qualcosa ancora non è chiaro.
 |
 |
|
|
Mrs.who
Nuovo Arrivato

13 Messaggi |
 Inserito il - 23 maggio 2017 : 22:34:47 Inserito il - 23 maggio 2017 : 22:34:47



|
Gentile Caffey ti ringrazio per avermi risposto, mi hai aiutata non poco :)
Ho appreso da poco le basi della citofluorimetria e non sono riuscita a trovare in nessuna fonte una spiegazione soddisfacente riguardo all'interpretazione di un grafico a plot come questo!
Se io ho inserito nel citofluorimetro una sospensione di sole piastrine (isolate precedentemente), i diversi colori (blu,cerde,rosso)fanno riferimento a diverse molecole antigeniche riconosciute sulla loro superficie cellulare?
In questo lavoro si valuta come cambia l'espressione dei marker di attivazione piastrinica in modelli sperimentali murini Germ Free e Specific Pathogen Free in seguito a trattamenti diversi (PCPA: mix di antibiotici; Sp: colonizzazione con Clostridi).
Ed il succo del lavoro è che il microbiota è fondamentale nella regolazione della funzionalità piastrinica perchè stimola l'espressione di questi marker.
Il probolema è che non riesco a capire come faccio a dedurre tutto ciò guardando questo grafico! Ti ringrazio ad ogni modo e ti auguro una buona serata Ti ringrazio ad ogni modo e ti auguro una buona serata  |
 |
|
|
Caffey
Utente Attivo
  

Città: Perugia

1496 Messaggi |
 Inserito il - 23 maggio 2017 : 23:06:38 Inserito il - 23 maggio 2017 : 23:06:38




|
Ciao.
Ora capisco meglio il problema. Intanto ti posso consigliare di fare questo breve e semplice corso introduttivo online: https://www.bdbiosciences.com/us/support/training/s/itf_launch.
Provo comunque a rispondere alle tue domande. In breve, nei plot citofluorimetrici puoi decidere cosa mettere in ascisse e cosa mettere in ordinate, in questo caso (ed è il plot base che si fa praticamente sempre per distinguere le popolazioni), hai dimensione dell'evento (nel tuo caso una piastrina) in ordinata e complessità intracellulare in ascissa. Ogni puntino rappresenta un evento ma quando sono tantissimi si sovrappongono e quindi è possibile, come in questo caso, utilizzare una rappresentazione "pseudocolor", che ti mostra in scala di colore la densità dei puntini presenti. Quindi i colori che vedi non rappresentano altro che la densità degli eventi: dove è rosso ci sono tantissimi puntini sovrapposti e progressivamente meno dove è arancio > giallo > verde > azzurro > blu.
Per capire il significato nel complesso, devi guardare i grafici a coppie, superiore ed inferiore. Il grafico superiore rappresenta le caratteristiche delle tue piastrine prese da quel modello non stimolate. Sotto, le loro caratteristiche dopo stimolazione con collagene (da quanto so è uno stimolo classico per indurre l'attivazionde delle piastrine). Dopo stimolazione, le piastrine aumentano la loro complessità "cellulare", preparandosi a svolgere le loro funzioni di secrezione e quant'altro (ripeto, non conosco la biologia delle piastrine e quindi intuisco e deduco dal grafico, potrei sbagliare ma non è importante ai fini della spiegazione). Questa variazione determina uno spostamento dell'evento verso destra, ovvero aumenta il valore di SSC.
Ora provo ad intepretare le prime due coppie di grafici, tu prova a fare il resto. Ci tengo a ribadire che la mia spiegazione biologica potrebbe non essere corretta, ma capisci che ho solo un'immagine di un articolo in un ambito che non conosco...
Abbiamo le piastrine prelevate da un modello SPF che, dopo stimolazione con collagene, per il 13,4% risultano attivate (contro un livello basale di 0,13%). Se trattiamo il modello con PCPA, le piastrine attive dopo stimolazione con collagene sono il 10,4% (basale dello 0,45%. Se guardi le altre 3 coppie, le differenze sono ancora più evidenti. Non posso tirare conclusioni, ovviamente!
Esistono molti marker di attivazione piastrinica che sicuramente gli autori avranno valutato utilizzando delle marcature fluorescenti, ma non si vedono in questi grafici.
 |
 |
|
|
Mrs.who
Nuovo Arrivato

13 Messaggi |
 Inserito il - 23 maggio 2017 : 23:30:06 Inserito il - 23 maggio 2017 : 23:30:06



|
Wow, finalmente una spiegazione come si deve che non dia per scontate le basi!!! 
Semplicemente grazie infinite per il tempo speso, spero che un giorno o l'altro potrò ricambiare la gentilezza! |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|






 Ti ringrazio ad ogni modo e ti auguro una buona serata
Ti ringrazio ad ogni modo e ti auguro una buona serata 
