| Autore |
Discussione |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 26 maggio 2009 : 10:27:29 Inserito il - 26 maggio 2009 : 10:27:29




|
Ciao a tutti,
sono una dottoranda in biologia cellulare e biotecnologie applicate agli organismi marini. Attualmente però mi sto occupando di biologia molecolare in Norvegia e non sono ancora molto ferrata in materia..
Ho un problemino...Ho provato a fare diversi tentativi di pcr provando a cambiare la temperatura di annealing e alla fine ho trovato delle bande della stessa lunghezza della proteina che sto cercando a 67.3°C, 64.1°C e a 60.3°C.
A questo punto ho cambiato il gradiente di temperatura (66.7-66.2-65.2-64-63-62.4-62) e ho trovato le bande in tutte le 8 repliche ma con una intensità diversa...Mi spiego meglio...Il tecnico con cui lavoro mi ha detto che dovrei trovare le bande con un segnale che aumenta con il diminuire della temperatura, ma l'intensità delle mie bande sembra non seguire questo criterio!!! Quale può essere il problema??? In cosa sbaglio???
Grazie a tutti!!!

|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 26 maggio 2009 : 10:47:27 Inserito il - 26 maggio 2009 : 10:47:27





|
Oh che bello qualcun altro al freddo nord!!! 
Benvenuta su MolecularLab! 
Citazione:
Il tecnico con cui lavoro mi ha detto che dovrei trovare le bande con un segnale che aumenta con il diminuire della temperatura, ma l'intensità delle mie bande sembra non seguire questo criterio!!!
In teoria a temperature più basse la reazione è meno stringente e potresti avere più amplificato, ma anche degli aspecifici.
A temperature più alte può essere che i primers non si appaiono bene e quindi avrai meno amplificato.
Però una volta che hai trovato una temperatura a cui funziona bene la tua PCR perché devi provare a varie temperature?
Selezionane una che va bene e utilizza quella.
Comunque dovresti specificare meglio:
Citazione:
l'intensità delle mie bande sembra non seguire questo criterio!!!
e com'è l'intensità delle tue bande in relazione alla temperatura?
Puoi mettere una foto o spiegare che intensità hai alle varie temperature? |
 |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 26 maggio 2009 : 11:04:48 Inserito il - 26 maggio 2009 : 11:04:48




|
grazie mille!!!
Sinceramente non so perchè mi fanno provare varie temperature...Ma qui tra problemi di comprensione e collaborazione è difficile avere spiegazioni chiare e plausibili...
Cmq ti allego la foto...Come vedi i primers non sono così specifici xche ho altre bande, ma ne ho ordinati altri e devo ancora provarli..Cmq hanno tutti la stessa temperatura..
L'andamento delle bande è casuale!!!
Immagine:
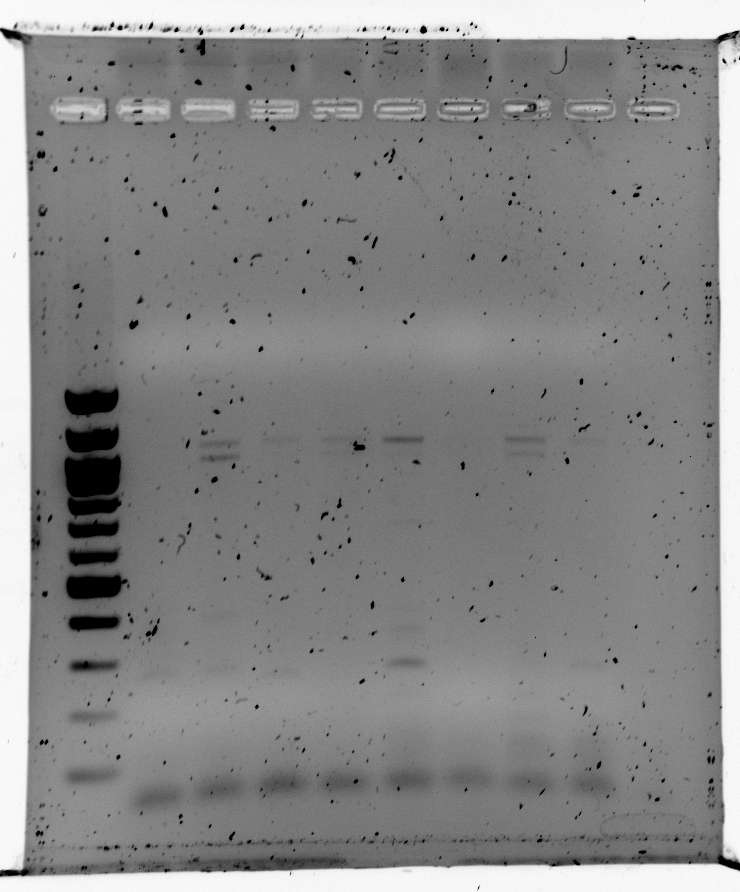
589,96 KB |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 26 maggio 2009 : 16:55:58 Inserito il - 26 maggio 2009 : 16:55:58





|
Beh oltre che avere la tua banda hai anche dei begli aspecifici, ma la tua banda esattamente quale sarebbe? La prima in alto?
Ma sei sicura che i primers siano abbastanza specifici e che non riconoscano anche altro?
Comunque vista l'immagine ti hanno detto di provare a varie temperature per cercare di eliminare l'aspecifico ed avere una banda migliore.
Però le bande aspecifiche che si vedono in basso le potresti anche eliminare, ma banda superiore vicino alla tua sembra veramente intensa!
Prova a controllare i primers, e visto che ne hai già ordinati altri prova a vedere con quelli cosa ottieni.

P.S.Citazione:
qui tra problemi di comprensione...
ah come ti capisco!!! |
 |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 26 maggio 2009 : 17:00:03 Inserito il - 26 maggio 2009 : 17:00:03




|
grazie mille!!!
Si ora provo gli altri primer!!!
 e buon finlandese!!! e buon finlandese!!!
PS:io mi rifiuto di imparare il norvegese!!! |
 |
|
|
max81
Nuovo Arrivato

2 Messaggi |
 Inserito il - 26 maggio 2009 : 22:25:53 Inserito il - 26 maggio 2009 : 22:25:53




|
hai provato a modificare la concentrazione del Mg2+ nella tua mix di PCR ? ovviamente puoi farlo se il buffer non viene già fornito con il Mg2+ all'interno.
il range da utilizzare può variare da 1 a 4 mM. per esperienza ti posso dire che la concentrazione giusta aiuta ad eliminare aspecifici. di solito i protocolli forniti ti dicono di usare 1,5 mM come standard. prova ad alzarlo ad intervalli di 0,5 usando la temperatura a cui ti sembra che l'amplificato sia buono. |
 |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 27 maggio 2009 : 10:00:15 Inserito il - 27 maggio 2009 : 10:00:15




|
grazie!

si io uso 1.5mM di mg2+ come da protocollo...ora provo a cambiare la concentrazione!
E poi vi dico!!!

PS: Nel frattempo mi hanno detto di provare con una sola temperatura (66°C)..Vediamo che succede!!! |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 27 maggio 2009 : 11:15:06 Inserito il - 27 maggio 2009 : 11:15:06





|
Si aumentare la concentrazione di Mg2+ rende più specifica la reazione! Ma se i risultati sono quelli della foto che hai postato, scusa ma sono un po' scettica!
Non ho capito bene quale delle 2 bande superiori sia quella giusta, ma sono molto simili come intensità e a volte viene fuori meglio una e a volte l'altra.
Io prima di tutto controllerei che la coppia di primers sia veramente specifica e non riconosca anche altro!
Hai provato a fare un blast? Puoi anche utilizzare Primer BLAST, metti la tua coppia di primers e faglieli controllare!
Inoltre da cosa amplifichi cDNA o DNA?
Citazione:
Messaggio inserito da max81
hai provato a modificare la concentrazione del Mg2+ nella tua mix di PCR ? ovviamente puoi farlo se il buffer non viene già fornito con il Mg2+ all'interno.
Aumentarla puoi comunque sempre, il problema si pone solo se vuoi diminuirla!
P.S. beh io non lo imparerò mai il Finlandese... ma qualche parolina mi è utile per cavarmela, almeno in labo!!!
  |
 |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 27 maggio 2009 : 12:07:41 Inserito il - 27 maggio 2009 : 12:07:41




|
Suppongo che la mia banda sia quella più bassa xche la lunghezza è 990 bp...
Purtroppo lavoro su pesci (sea bass appunto!!) e in rete non c'è quasi nulla...primer blast mi dice che i primer non sono specifici... Proverò gli altri che ho ordinato se anche la pcr che sto facendo ora non viene!!! Proverò gli altri che ho ordinato se anche la pcr che sto facendo ora non viene!!!
Amplifico da cDNA...
Cmq grazie veramente tanto x tutti i consigli!!!Mi fate quasi sentire a casa!! |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 27 maggio 2009 : 13:01:59 Inserito il - 27 maggio 2009 : 13:01:59





|
Sui i pesci mi trovi proprio impreparata!
So solo che dalle nostre parti ci possiamo fare delle belle scorpacciate di salmone! 
Comunque se non sono specifici le altre bande sono dovute a quello.
La cosa migliore sarebbe cercare di avere una coppia di primers più specifici.
Puoi usare Primer Blast anche per disegnare primers specifici.
(se la sequenza della tua specie è inserita nel database!)
Ti chiedevo se era da cDNA perché potrebbe anche essere che la banda più alta sia DNA (anche se differenza di grandezza è veramente poca), dipende da come sono i primers e se possono amplificare anche DNA. Fai trattamento con DNasi dei tuoi campioni?
In ogni caso è meglio disegnare i primers a cavallo degli esoni così si elimina questo problema.
  |
 |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 27 maggio 2009 : 13:40:25 Inserito il - 27 maggio 2009 : 13:40:25




|
lo so ma purtroppo la sequenza non è nel database  ... ...
Non faccio nessun trattamento con DNasi..dovrei? 
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 27 maggio 2009 : 14:37:25 Inserito il - 27 maggio 2009 : 14:37:25





|
Citazione:
Messaggio inserito da seabass
lo so ma purtroppo la sequenza non è nel database  ... ...
eh questo è un problema purtroppo!!!
Ma i primers che hai come sono stati disegnati? Su che sequenza sono stati scelti?
Citazione:
Non faccio nessun trattamento con DNasi..dovrei? 
beh in genere è raccomandato farlo, ti evita la contaminazione da DNA genomico! |
 |
|
|
seabass
Nuovo Arrivato

25 Messaggi |
 Inserito il - 27 maggio 2009 : 15:01:29 Inserito il - 27 maggio 2009 : 15:01:29




|
Su una sequenza di cDNA incompleta nella stessa specie che sto studiando!!!!
Magari provo con la DNasi...
 |
 |
|
| |
Discussione |
|








