| Autore |
Discussione |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 13 settembre 2009 : 19:05:43 Inserito il - 13 settembre 2009 : 19:05:43





|
Qualcuno sa come posso fare per capire se un determinato
fluorocromo (nel mio caso: phycoerythrin, PE) fa spectral
overlap con un altro di colore simile (mCherry) ?
Vorrei capire se è possibile usarli assieme
per un'analisi citofluorimetrica OPPURE per una
co-immunofluorescenza su vetrino (riuscendo a
distinguere le singole marcatura dalla doppia marcatura)
|
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
|
|
|
|
Gabriele
Utente
 
Prov.: Pisa
Città: Pisa

615 Messaggi |
 Inserito il - 13 settembre 2009 : 20:06:21 Inserito il - 13 settembre 2009 : 20:06:21




|
| Credo che soltanto il confronto degli spettri di assorbimento e di emissione possa chiarire la questione. |
"Chi rinuncia alla libertà per la sicurezza, non merita né la libertà né la sicurezza. E finirà col perdere entrambe." |
 |
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 13 settembre 2009 : 20:12:33 Inserito il - 13 settembre 2009 : 20:12:33




|
concordo,
aggiungerei che la ricerca di qualche riferimento bibbliografico non guasterebbe...
ti consiglierei inoltre di chiamare la ditta che li vende e di chiedere a loro
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 13 settembre 2009 : 22:22:19 Inserito il - 13 settembre 2009 : 22:22:19




|
| Ricorda che anche se gli spettri non si sovrappongono puoi comunque avere color bleeding. Dipende non solo dagli spettri, ma dai filtri che usi, dall'intensità della fluorescenza (e questo è molto importante nel tuo caso visto che probabilmente non potrai controllare il livello di espressione di mCherry), dalla potenza della lampada/laser che usi etc. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 14 settembre 2009 : 01:24:02 Inserito il - 14 settembre 2009 : 01:24:02





|
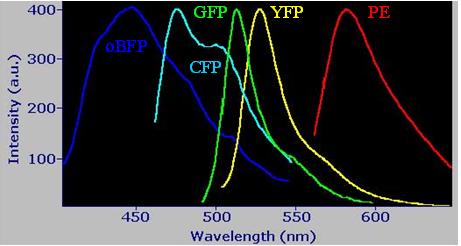
Urca quante cose. Io mi annoto tutto: in fondo ho un anno per imparare.
Solo un'ultima domanda: guardando lo spettro riportato da GFPina sembra
che la CFP e la GFP abbiano un overlap considerevole, EPPURE pare che in
diversi lavori (tra cui uno che conosco bene) sia stata usata una doppia
marcatura CFP + GFP senza problemi di compensazione... c'è una spiegazione
semplice/ovvia che mi sfugge?? |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 14 settembre 2009 : 09:19:12 Inserito il - 14 settembre 2009 : 09:19:12




|
Ci sono metodi di spectral deconvolution che ti permettono di separare ad es. CFP, GFP e YFP nello stesso campione.
Ad es. puoi usare il sistema META di Zeiss (ce ne sono anche altri).
In alternativa si possono usare opportune varianti delle xFP e combinazioni particolari di filtri di emissione / eccitazione. Inoltre è molto importante ricordardi che hai un aumento di bleedthrough se fai un capture dei 2 fluorofori allo stesso tempo invece di farlo in sequenza. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 14 settembre 2009 : 16:00:04 Inserito il - 14 settembre 2009 : 16:00:04





|
Dunque, considerando che io sono un novizio assoluto e non saprei
pianificare analisi di quel genere neppure in linea puramente teorica
(non faccio ancora l'esperimento in pratica) che ne direste a riguardo
della 'feasibility' di un'analisi FACS multipla in cellule esprimenti
contemporaneamente tutti e tre questi reporter genes:
> dGFP (destabilized green fluorescent protein)
> deltaNGFR (-> Ab PE-coniugato)
> optimized BFP (blue fluorescent protein)
(ho rinunciato alla mCherry)
Immagine:
19,92 KB
In letteratura non ho trovato riscontri |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 14 settembre 2009 : 23:38:22 Inserito il - 14 settembre 2009 : 23:38:22





|
Ok, thanks. Immagino che ci voglia uno strumento di ultima generazione tipo questo.  |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 15 settembre 2009 : 08:13:20 Inserito il - 15 settembre 2009 : 08:13:20




|
| Dimenticavo di dire: non ho mai usato un FACS in vita mia... però alla fine che tu stia usando un confocale (quello l'ho usato!) o un FACS il principio della fluorescenza sempre quello è! |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
| |
Discussione |
|






