| Autore |
Discussione |
|
|
nina78
Nuovo Arrivato

3 Messaggi |
 Inserito il - 30 novembre 2010 : 21:40:17 Inserito il - 30 novembre 2010 : 21:40:17




|
Ciao a tutti!
sto studiando per l'esame di biologia molecolare..non riesco a capire come fare ad inserire ad esempio URA al posto di un gene X...
Grazie mille!!
|
|
|
|
|
Patrizio
Moderatore
  

Cittā: Barcellona

1915 Messaggi |
 Inserito il - 01 dicembre 2010 : 10:27:56 Inserito il - 01 dicembre 2010 : 10:27:56





|
Dove lo devi inserire X ? genoma...plasmide ... frammento ... sii piu' chiara
Benvenuta su molecularlab |

|
 |
|
|
nina78
Nuovo Arrivato

3 Messaggi |
 Inserito il - 01 dicembre 2010 : 11:46:28 Inserito il - 01 dicembre 2010 : 11:46:28




|
ciao Patrizio!
lo devo mettere all'interno del genoma...
grazie mille! |
 |
|
|
Patrizio
Moderatore
  

Cittā: Barcellona

1915 Messaggi |
 Inserito il - 01 dicembre 2010 : 12:16:08 Inserito il - 01 dicembre 2010 : 12:16:08





|
| quale essere vivente? |

|
 |
|
|
nina78
Nuovo Arrivato

3 Messaggi |
 Inserito il - 01 dicembre 2010 : 12:24:38 Inserito il - 01 dicembre 2010 : 12:24:38




|
| nel cromosoma 2 di S. cerevisiae devo introdurre il marcatore triptofano nel locus per ADE2..scusa se non sono stata chiara! |
 |
|
|
Patrizio
Moderatore
  

Cittā: Barcellona

1915 Messaggi |
 Inserito il - 01 dicembre 2010 : 12:57:07 Inserito il - 01 dicembre 2010 : 12:57:07





|
| ..non so a cosa ti serva inserire un marcatore al posto di un altro marcatore (se ho capito bene), tuttavia devi cotruire un frammento formato dal tuo gene TRP che fiancheggia delle sequenze omologhe alle regioni 5' e 3' di ADE2 in questa maniera si ha ricombinazione omologa e potrai selezionare i positivi in un terreno minimo ADE2+ TRP- , ovviamente usando ceppi che siano nel loro gene wild type TRP- |

|
 |
|
|
Patrizio
Moderatore
  

Cittā: Barcellona

1915 Messaggi |
 Inserito il - 01 dicembre 2010 : 13:03:14 Inserito il - 01 dicembre 2010 : 13:03:14





|
Immagine:
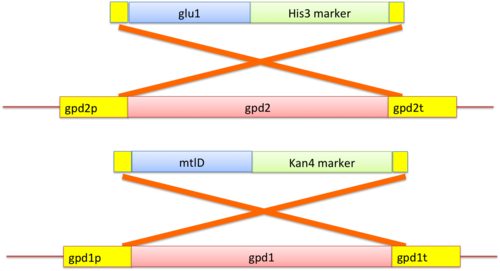
36,46 KB |

|
 |
|
|
Patrizio
Moderatore
  

Cittā: Barcellona

1915 Messaggi |
 Inserito il - 01 dicembre 2010 : 13:04:32 Inserito il - 01 dicembre 2010 : 13:04:32





|
| questo sopra puo' essere un esempio , sono uguali con diversi geni |

|
 |
|
|
ilabest
Nuovo Arrivato

8 Messaggi |
 Inserito il - 23 maggio 2011 : 20:45:10 Inserito il - 23 maggio 2011 : 20:45:10




|
Ciao a tutti! Avrei bisogno di sapere come funzionano le cassette di inattivazione con marcatore di selezione.. prendendo in considerazione S.cerevisiae come organismo, come faccio? Trasformo con un plasmide? Esempio: per inattivare il mio gene X, uso una cassetta di inattivazione con il marcatore di selezione Ura: i ceppi che crescono in assenza di Ura, saranno quelli che presentano il mio gene X inattivo? Non capisco bene il meccanismo alla base...
Grazie mille!! |
 |
|
|
thejoint84
Nuovo Arrivato

46 Messaggi |
 Inserito il - 23 maggio 2011 : 21:27:47 Inserito il - 23 maggio 2011 : 21:27:47




|
| Allora... Tu vuoi inattivare il tuo gene X in S.Cerevisiae... Usi come Patrizio ha spiegato prima nel post un plasmide che ha il tuo marcatore URA fiancheggiato ta regioni omologhe (uguali) a quelle del gene che vuoi inattivare. E' importantissimo che tu usi un ceppo URA-, perchč sennō non potrai mai selezionare i lieviti che hanno subito la ricombinazione..... Quello che succede č che il tuo gene X viene sostituito da URA, perdi la funzione di X, ma in cambio puoi sintetizzare uracile..... e il processo si chiama ricombinazione omologa.... |
http://cornermolecularbiology.blogspot.com/
|
 |
|
|
0barra1
Utente Senior
   

Cittā: Paris, VIIčme arrondissement

3847 Messaggi |
 Inserito il - 23 maggio 2011 : 21:31:55 Inserito il - 23 maggio 2011 : 21:31:55




|
| Ciao, puoi trasdurre S. cerevisiae con un plasmide contenente il marcatore desiderato fiancheggiato da sequenze del gene da inattivare. Per ricombinazione omologa il tuo costrutto plasmidico andrā ad integrarsi nel cromosoma, excidendo il gene target. |
So, forget Jesus. The stars died so that you could be here today.
A Universe From Nothing, Lawrence Krauss
|
 |
|
|
ilabest
Nuovo Arrivato

8 Messaggi |
 Inserito il - 24 maggio 2011 : 13:06:12 Inserito il - 24 maggio 2011 : 13:06:12




|
Grazie mille per le risposte!! Ora ho capito 
 |
 |
|
| |
Discussione |
|








