Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
Shatzy
Nuovo Arrivato
Città: PEsaro

6 Messaggi |
 Inserito il - 17 gennaio 2008 : 17:17:09 Inserito il - 17 gennaio 2008 : 17:17:09





|
Salve a tutti, avrei una questione da porvi, riguardo a un'alterata moibilità elettroforetica di alcuni miei campioni di RNA.
In alcuni casi, ho notato che le due subunità ribosomiali, corrono nei gel d'agaroso, in modo diverso dal solito, ovvero la subunità maggiore resta più in alto e la minore più in basso (quindi non ho uno shift vero e prorpio, non sono tutte e due pià in alto o più in basso) addirittura in alcuni casi ho delle bande "doppie" dove le interne sono quelle "standard", e le esterne, sono queste delle quali vi parlo. Siccome alla presenza delle sole bande "esterne" ho problemi nell'amplificazione del cDNA ottenuto da tale RNA (non sono problemi dovuti alla presenza di inbitori), mi chiedevo se avete qualche idea riguardo a cosa possa alterare la mobilità elettroforetica dell'RNA. Io credo che questo qualcosa, possa interferire con la sintesi del cDNA non apportando degli inibitori, ma rendendo meno "disponibile" l'RNA e meno effeiciente la retrotrascrizione...boooooh?!???
Intanto grazie...ho paura di non essere stata molto chiara..
|
|
|
|
|
cin
Utente Junior


Prov.: Padova
Città: Padova

558 Messaggi |
 Inserito il - 18 gennaio 2008 : 13:38:51 Inserito il - 18 gennaio 2008 : 13:38:51




|
In effetti non è che c'ho capito gran che. Parli di mobilità elettroforetica intendendo la corsa all'Agilent?
Se tu riuscissi ad incollare qui le immagini della corsa magari potremmo discuterne tutti insieme e darti dei suggerimenti.
Forse sarebbe meglio che ci esponessi dall'inizio su cosa lavori ( cellule, tessuti...) e quale sono gli inghippi che incontri.
Parli di problemi di corsa nel gel d'agarosio, ma anche di problemi nell'amplificazione, potresti essere più dettagliata possibile? |
 |
|
|
Shatzy
Nuovo Arrivato
Città: PEsaro

6 Messaggi |
 Inserito il - 18 gennaio 2008 : 16:07:24 Inserito il - 18 gennaio 2008 : 16:07:24





|
Effettivemente...allora faccio studi di espresione genica su alghe unicellulari, usando come tecnica il Differential Display, quindi, in breve, estraggo l'RNA dalle alghe quando si trovano in due differenti stati cellulari (quello in fase vegetativa, cioè quando sono attive e si dividono per semplice mitosi, e quello di cisti, fase di resistenza ottenuta dall'incrocio di due ceppi, un + e un -).
L'RNA messaggero viene poi diviso in tre subpopolazioni sulla base dell'ultimo nucleotide prima della coda di PolyA (usando degli oligo dT che si chiamano ancorati) e infine ogni sub popolazione viene amplificata, una alla volta, in una PCR con random primers. I prodotti della PCR si fanno correre su un gel di acrilammide, sulla base della corsa elettroforetica si confrontano le differenze fra i due stati cellulari, si isolano le bande che corrispondono a geni differenzialmente espressi, si seqenziano e clonano e infine si validano i risultati su nuove preparazioni di RNA.
Fatta questa premessa, io, quando estraggo l'RNA, faccio generalmente un gel d'agaroso, in condizioni non denaturanti, che faccio correre molto velocemente giusto per vedere "che faccia ha" l'RNA.
Nel caso dell'estrazione dell'RNA di cisti, il primo gel fatto aveva quasta faccia qui:
Immagine:
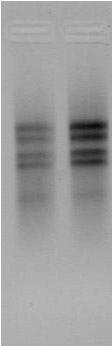
25,06 KB
le bande erano doppie!! Io al tempo diedi la colpa ai due differenti contributi dei deu mating type, ma effettivemnte...non ha molto senso che l'RNA ribosomiale (che è in pratica quello che si vede sul gel) sia diverso! Fra l'altro i 2 ceppi usati per l'incrocio, hanno il profilo elettroforetico "classico" con le due bande e le due bande, sembrano essere le due bande "interne" fra le 4 che vedete in questo strano gel (P.S. non è un artefatto della corsa eprché è riproducibile).
Io ho usato comunque quell'RNA per le mie prove e ho identificato dei geni potenzialmente differenzialmente espressi. Al momento della validazione però, non ho più ritrovato quei geni nelle cisti (e, fra l'altor, nelle cisti dovevano essere over-espressi) vado a fare i gel e vedo che:
a) il profilo elttroforetico non ha più dato quelle doppie bande, ma due semplici bande, una per ciascuna subunità ribosomiale
b) le due bande presenti sono le due bande "esterne"
Questo dato, mi è stato confermato anche da una ragazza tedesca che collabora con noi, lei ha usato il BioAnalyzer dell'agilent e ha ritrovato queste stesse cose e, per di più, ha anche avuto il doppio picco. Questo è il profilo dell'agilent, mi riferisco ai campioni 5,6, e 7, che hanno solo le bande "esterne" e al campione 8 che ha il doppio bandeggio:
Immagine:
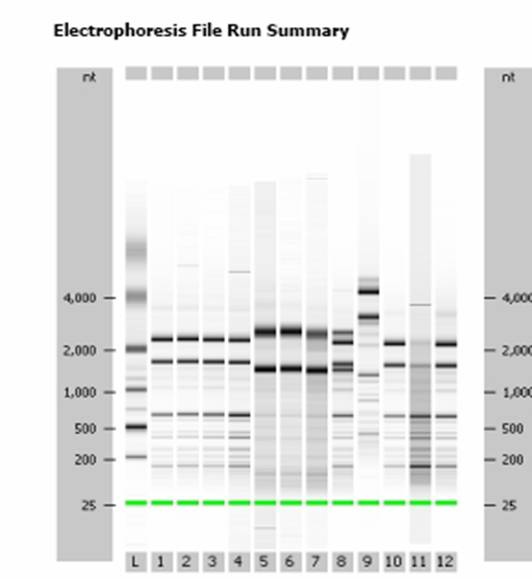
20,43 KB
Il mio dubbio a questo punto è: cosa altera in quel modo la mobilità elettroforetica delle due subunità ribosomiali? Cosa può essere? E' di sicuro un qualcoas che non inibisce la PCR (ho fatto le prove con del DNA plamidico), ma che forse mi rende meno disponibile l'RNA al momento della sintesi del cDNA (che io non doso mai). In pratica pensavo a interazion RNA-RNA o a RNA interference, però se il BioAnalyzer lavora in condizioni denaturanti...tutta la mia pseudo teoria cade!!
spero stavolta di essere stata un po' più chiara...è che è proprio la questione che è un po' complicata e io no nso più che pesci pigliare!!!
Grazie a tutti! |
 |
|
|
cin
Utente Junior


Prov.: Padova
Città: Padova

558 Messaggi |
 Inserito il - 21 gennaio 2008 : 12:07:33 Inserito il - 21 gennaio 2008 : 12:07:33




|
Sinceramente non ho nessuna esperienza nel campo da te trattato, per cui anche se sei stata molto chiara faccio difficoltà a seguirti e a darti qualche aiuto.
Avrò fatto un centinaio di corse con l'Agilent, ma non ho mai visto profili come quelli del campione 8 (nè tanto meno come i camp da 1 a 4 e da 9 a 12, in genere i nostri sono come 5,6,7).
Avendo quelle bande "strane" sull'RNA, a me verrebbe da pensare ad un problema insito nell'estrazione o campione dipendente. Non sapendo nulla di alghe e del loro RNA ti consiglierei di chiedere un apporto tecnico agli specialist della Applied Biosystems, che da quando hanno assorbito anche l'Ambion (specialista per ciò che riguarda l'RNA) direi che siano quelli più competenti in materia ( nessuno me ne voglia).
Sempre che qualcuno... tipo GFPina non ti dia una mano. |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|






