Per mantenere il testo allineato devi utilizzare il tag code:
[code ] [ /code]
quello che scrivi in mezzo rimarrà allineato.
Vedi qua per l'utilizzo dei vari tag: Come formattare il testo
Allora per quanto riguarda la risposta io intendevo una cosa fatta così:
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
dove in rosso sono gli adattatori e in blu il frammento, come li hai disegnati tu gli adattatori avresti un filamento blut che dovresti tagliare con PstI per renderli blunt.
Ma non continuerei su questa strada, scusa ho sbagliato io prima!
Una strategia più intelligente è quella di fare un adapter che si va a legare al vettore, (come farebbe il frammento con i siti giusti per intenderci) quindi conterrà all'esterno le sequenze complementari a quelle del vettore (tagliato da PstI) e all'interno la sequenza di XhoI, il vettore si richiuderà, poi tagli con XhoI e hai i siti complementari a quelli del tuo vettore.
La strategia è quella che vedi in figura:
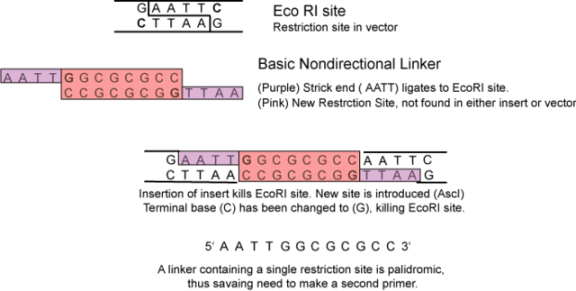
In questo modo ti costruisci l'adattatore, che sarà una sequenza a singolo filamento (indicata da 5' a 3'), palindroma che si appaia con sé stessa facendo l'adattatore.
Questo è in effetti quello che ti chiedeva l'esercizio.
A questo punto prova a scrivere la sequenza, che è anche più facile rispetto a prima!







