Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
daisy
Nuovo Arrivato
Prov.: Asti
Città: asti

108 Messaggi |
 Inserito il - 05 gennaio 2008 : 21:07:09 Inserito il - 05 gennaio 2008 : 21:07:09




|
scusate se vi rompo ancora con la genetica , ma sono proprio disperata!!! Ho avuto una ricaduta con l'influenza adesso, ho la febbre a 39° , ma stoica cerco(almeno ci provo, con scarsi risultati ) di preparare alcuni esercizi di genetica. Ma su due sono in profonda crisi 1) Una femmina di Drosophila con genotipo AaBb viene incrociata con un maschio doppio recessivo aa bb . La loro progenie include 442 AaBb, 458 aabb, 46 Aabb e 54 aaBb. Spiegate questi risultati . Quale coppie di loci sono concatenate fra loro (sempre se c'è concatenazione) ? Non credo che ci sia segregazione indipendente come quella mendeliana , ma piuttosto una meiosi, o anche più meiosi ma non so che fare. Come lo imposto se nella maniera classica, ovvero quattro gameti ottenuti dal genotipo eterozigote e uno dall' omozigote recessivo? 2)Un moscerino da frutta BR/br è incrociatocon un doppio recessivo br/br. Nell'84% delle meiosi nessun chiasma avviene tra le coppie di geni concatenati , nel 16 %delle meiosi avviene la formazione di un chiasma tra questi due loci. Quale % della progenie sarà Bbrr? le alternative sono 4%,16%,84% e 16%. non capisco i geni concatenati sono B e R ho sto prendendo una cantonata sono confusa sempre più confusa . C'è qualche anima santa che protrebbe aiutarmi non impongo nè immediatamente, nè alle calende greche, entro martedì della prossima settimana , se un vostro prezioso aiuto giungerà prima ben venga. speriamo che l'influenza mi abbandoni , se no dovrò tornare all'ospedale , mi succede un anno sì e l'altro pure che palle ... ciao e grazie comunque 
|
e@daisy |
|
|
|
|
imy85x
Nuovo Arrivato

Città: napoli

110 Messaggi |
 Inserito il - 06 gennaio 2008 : 10:51:44 Inserito il - 06 gennaio 2008 : 10:51:44




|
Allora:
Esercizio 1):
Devi inizialmente ipotizzare che avvenga segregazione indipendente secondo la teoria mendeliana,in tal caso il rapporto fenotipico atteso è di 1:1:1:1
quindi nel tuo caso i risultati del tuo incrocio dovrebbero dare rapporto fenotipico di:
250:250:250:250
poichè i risultati si discostano da tale ipotesi perchè il numero dei parentali AaBb e aabb è maggiore rispetto ai ricombinanti aaBb e Aabb devi dedurre che i tuoi geni A e B siano concatenati.
A Tale deduzione ci puoi arrivare anche effettuando il test del X2(chi-quadro)e da tale test risulta che la probabilita che la deviazione dei valori osservati rispetto a quelli attesi sia dovuta al caso è <<(molto minore) di 0.05
quindi l'ipotesi che i 2 geni siano indipendenti è da scartare.
Di conseguenza i 2 geni sono concatenati.
Per calcolare la distanza di mappa dei due geni dovresti calcolare la frequenza di crossing-over tra questa regione del cromosoma durante la meiosi,poichè ciò non si può fare diretamente,calcoliamo la frequenza dei ricombinanti che deriva da tali crossing-over.
Essa è uguale a :
n°dei ricombinanti/progenie totale
nel tuo esercizio è :
(46+54)/1000 x100 = 10%
Dato che la frequenza di ricombinazione può essere utilizzata per determinare la distanza di mappa,specialmente se la distanza è piccola,possiamo concludere che la distanza tra A e B è di 10um(unità di mappa)
Esercizio 2)
Per il secondo esercizio...sono molto perplessa..
onestamente credevo che,dato il 16% di ricombinanti ed l'84%di parentali,la %di Bbrr fosse stata 8%...poichè pensavo che 16% era il risultato di: 8%Bbrr e 8% bbRr
Però 8% non è presente tra le opzioni...probabilmente è 4% poichè c'è un fenomeno di interferenza dato che il numero dei ricombinanti atteso (8%) è minore del numero dei ricombinanti osservati(4%)
Beh..sul primo sono sicura.. |
Imy
Le persone che meritano fiducia sono quelle che ti stanno accanto anche quando non le ami abbastanza.
|
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 06 gennaio 2008 : 19:21:35 Inserito il - 06 gennaio 2008 : 19:21:35





|
Sono d'accordissimo sul primo esercizio!
Per il secondo invece devi tener presente che:
"il Crossing-Over avviene allo stadio di 4 filamenti ma ne coinvolge solo 2!" (frasetta magica detta dalla mia prof di genetica che dopo 10 anni ricordo ancora  ) )
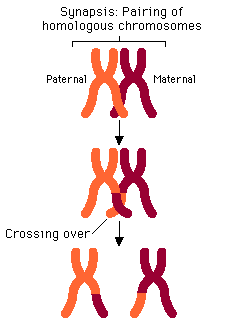
Quindi nel tuo caso:
- nel 16% delle meiosi si ha crossing-over:
B R B r
____________ --> ____________
b r b R
- ma solo la metà dei cromatidi sono coinvolti (2 su 4!) quindi hai l'8% di ricombinazione!
- quindi avrai 4% di Br e 4% di bR
  |
 |
|
|
imy85x
Nuovo Arrivato

Città: napoli

110 Messaggi |
 Inserito il - 06 gennaio 2008 : 19:47:29 Inserito il - 06 gennaio 2008 : 19:47:29




|
E' verooo...eheheheh mi sono persa in un bicchier d'acqua!!
   |
Imy
Le persone che meritano fiducia sono quelle che ti stanno accanto anche quando non le ami abbastanza.
|
 |
|
|
fish89
Nuovo Arrivato

5 Messaggi |
 Inserito il - 10 marzo 2012 : 11:19:53 Inserito il - 10 marzo 2012 : 11:19:53




|
| Ciao GFPina, leggendo la tua risposta mi è venuto un dubbio. Penso di aver capito il perchè con 4 filamenti (la coppia di omologi) solo due partecipano al crossing over. Ma se invece di 2 crossing over avessi 4 crossing over, i filamenti impegnati non sono più 2 ma 4?Grazie in anticipo a presto |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 10 marzo 2012 : 15:34:12 Inserito il - 10 marzo 2012 : 15:34:12





|
Citazione:
Messaggio inserito da fish89
Ciao GFPina, leggendo la tua risposta mi è venuto un dubbio. Penso di aver capito il perchè con 4 filamenti (la coppia di omologi) solo due partecipano al crossing over. Ma se invece di 2 crossing over avessi 4 crossing over, i filamenti impegnati non sono più 2 ma 4?Grazie in anticipo a presto
Ogni "singolo" crossing-over coinvolge 2 filamenti appartenenti ciascuno ad un cromosoma omologo diverso.
Quindi se chiamiamo 1 e 2 i filamenti di un omologo e 3 e 4 i filamenti dell'altro omologo (non sto a farti disegni) potrai avere c.o. tra: 1-3 o 1-4 o 2-3 o 2-4. Sono in ogni caso coinvolti solo 2 filamenti alla volta.
Se hai un doppio crossing over ad ogni evento, cioè per ognuno dei due c.o. avrai queste 4 possibilità.
Quindi ammettiamo per es. che il primo c.o. sia avvenuto tra 1-3, al secondo avrai ancora le quattro possibilità quindi il risultato sarà:
- 1-3 --> sono stati coinvolti in tutto solo due filamenti (sempre 1 e 3)
- 1-4 --> sono stati coinvolti inl tutto tre filamenti (1, 3 e 4)
- 2-3 --> sono stati coinvolti in tutto tre filamenti (1, 2 e 3)
- 2-4 --> sono stati coinvolti tutti e quattro i filamenti (1, 2, 3 e 4)
Non so perchè tu mi abbia chiesto con 4 c.o. rispetto a 2, forse ti sei confuso, comunque con più c.o. la cosa è analoga, ad ogni c.o. avviene una delle 4 possibilità. |
 |
|
|
fish89
Nuovo Arrivato

5 Messaggi |
 Inserito il - 10 marzo 2012 : 16:40:19 Inserito il - 10 marzo 2012 : 16:40:19




|
| Si si mi son confuso, intendevo 2 crossing over e non 4. Ma se un doppio crossing over coinvolgesse tutti e 4 i filamenti, quindi io avrei tutti e 4 i gameti ricombinanti giusto? e non solo i 2/4 dei gameti ricombinanti. Mi sa che sto facendo un bel po' di confusione. Scusa!!!! cmq ti ringrazio per la tua immediata disponibilità, se puoi rispondermi di nuovo ne sarei molto grato. a presto |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 11 marzo 2012 : 11:02:28 Inserito il - 11 marzo 2012 : 11:02:28





|
Allora sul fatto che per un singolo c.o. abbiamo metà parentali e metà ricombinanti perché vengono coinvolti solo due filamenti su 4, e il risultato è lo stesso qualunque siano i due filamenti (1-3, 1-4, 2-3 o 2-4) ci siamo vero? Questa è la base.
Ora come ti dicevo prima, il secondo c.o. può coinvolgere (come per il primo) qualunque dei 4 filamenti, purché siano ciascuno di uno dei due omologhi. Quindi ancora: 1-3, 1-4, 2-3 o 2-4, questi eventi sono equibrobabili. (questa è la cosa fondamentale per capire tutto il resto)
Ora è vero come dicevi tu che se hai un doppio c.o. che coinvolge tutti e 4 i filamenti ottieni "tutti ricombinanti", ma se ci pensi è anche vero che invece se il doppio crossing over coinvolge sempre gli stessi due filamenti ottieni "tutti parentali".
Riassumendo le 4 possibilità sono:
- doppio c.o. a 2 filamenti: 4 parentali
- doppio c.o. a 3 filamenti: 2 parentali e 2 ricombinanti
- doppio c.o. a 3 filamenti: 2 parentali e 2 ricombinanti
- doppio c.o. a 4 filamenti: 4 ricombinanti
La media dà: 50% parentali e 50% ricombinanti.
Visto che non ho voglia però di mettermi a fare disegni, per le immagini ti rimando a questo link, dove è anche in parte spiegato quello che ho detto:
http://www.ag.ndsu.nodak.edu/plantsci/adv_genetics/genetics/iodc/iodc02.htm |
 |
|
|
fish89
Nuovo Arrivato

5 Messaggi |
 Inserito il - 11 marzo 2012 : 13:55:22 Inserito il - 11 marzo 2012 : 13:55:22




|
| Si si perfetto!!!!Sei stata chiarissima!!!!!! non so come ringraziarti!!!!!grazie grazie grazie grazie!!!!! |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|








