| Autore |
Discussione |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 07 ottobre 2012 : 18:02:00 Inserito il - 07 ottobre 2012 : 18:02:00




|
eccomi con una nuova domanda!! qui in realtà spero di aver dato la risposta giusta! confermate per favore?
Hai del DNA in provetta: come discrimini che sia umano e di che sesso è?
1)io penso che la cosa più semplice da fare sia fare uno studio di finger printing, trovare STR, che sono specifiche per l'uomo.
però ho pensato anche che potrei cercare anche le sequenze ALU. se non sbaglio sono specifiche anche quelle?
2) sulla parte del sesso sono sicura: faccio analisi per gene per amelogenina, un gene che sul cromosoma X ha sequenza lunga 106bp e sul cromosoma Y è 112 bp. quindi se l'ibridazione e successiva elettroforesi mi darà una sola banda sarà XX e quindi femmina, se ho eterozigote, due bande, allora è XY cioè maschietto!!!!
potrei fare il cariotipo, ma ho escluso questa ipotesi perchè il cariotipo si fa estraendo DNA da cellule in meiosi, e il testo questo non lo dice, quindi non posso..... giusto???
grazie!!!
|
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
|
|
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 07 ottobre 2012 : 21:07:21 Inserito il - 07 ottobre 2012 : 21:07:21





|
Per quanto riguarda la prima direi le sequenze ALU che sono presenti solo nei primati. Poi in effetti si possono andare a vedere anche STR, esistono STR specifici per ogni specie. Sono utilizzati anche per distinguiere diversi individui all'interno della stessa specie.
Per la seconda direi entrambe quella suggerite, amelogenina o SRY però personalmente darei una prevelenza alla amelogenina. Tra l'altro l'amelogenina è quella inserita tra i marcatori del CODIS (Combined DNA Index System). L'analisi dell'amelogenina ti permette anche di analizzare campioni misti perchè puoi quantificare il rapporto X:Y, se hai ad es. un rapporto 3:1 sarai in presenza di un DNA misto maschile e femminile.
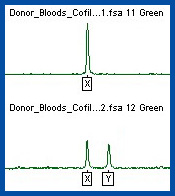
@ chick80: non è affatto scomodo andare a vedere la differenza di 6bp, viene fatto in genere con elettroforesi capillare e visualizzando un'elettroferogramma in cui vedi due picchi distinti per X e Y. Un campione maschile avrà due picchi mentre uno femminile avrà un picco solo di altezza doppia.
 |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 08 ottobre 2012 : 07:47:44 Inserito il - 08 ottobre 2012 : 07:47:44




|
Citazione:
@ chick80: non è affatto scomodo andare a vedere la differenza di 6bp, viene fatto in genere con elettroforesi capillare e visualizzando un'elettroferogramma in cui vedi due picchi distinti per X e Y. Un campione maschile avrà due picchi mentre uno femminile avrà un picco solo di altezza doppia.
Ragionavo pensando a farlo nella realtà. E nella realtà non posso fare un'elettroforesi capillare nel mio lab   |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 08 ottobre 2012 : 09:01:36 Inserito il - 08 ottobre 2012 : 09:01:36




|
grazie... mi era stato detto di farlo con gli STR, ma io ero convinta che fosse meglio farlo con gli ALU.
l'amelogenina penso sia più appropriato solo perchè è l'unico che ho tra gli appunti, quindi lo ha detto il docente... meglio dar importanza a questa tecnica!!!
grazie 1000 ad entrambi! |
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
 |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 08 ottobre 2012 : 09:29:27 Inserito il - 08 ottobre 2012 : 09:29:27




|
ovviamente per la prima domanda, faccio PCR, ibridazione con sonda ALU e poi elettroforesi.... tutto quello che mi interessa è solo vedere se la sonda ha ibridato, giusto?
|
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 08 ottobre 2012 : 20:52:27 Inserito il - 08 ottobre 2012 : 20:52:27





|
Citazione:
Messaggio inserito da chick80
Ragionavo pensando a farlo nella realtà. E nella realtà non posso fare un'elettroforesi capillare nel mio lab  
Va beh ok allora nella realtà io se dovessi discriminare se è DNA umano o animale, visto che al massimo potrebbe succedermi di avere una provetta con DNA umano o di topo, non farei niente di quello detto ma utilizzerei primers che ho già che amplificano sequenze umane o di topo e ci farei una PCR.
Ma non sarebbe questo il senso della domanda di prossima biologa! 
P.S. comunque al massimo potresti fare un gel di poliacrilammide e vedresti la differenza di 6bp! 
prossima biologa ti consiglio di rivedere questa cosa:
Citazione:
Messaggio inserito da prossima biologa
ovviamente per la prima domanda, faccio PCR, ibridazione con sonda ALU e poi elettroforesi....
forse hai un po' di confusione sulle tecniche. |
 |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 09 ottobre 2012 : 19:25:13 Inserito il - 09 ottobre 2012 : 19:25:13




|
un pò di confusione??? mi hai trattato con i guanti...
non so se quello che dico ora è corretto, perchè come hai detto tu, ho dei problemi a comprendere le tecniche...
comunque mi sono resa conto in questo istante leggendo che ho confuso proprio l'ordine dei passaggi, speriamo che ora non commetto errori:
ho DNA in provetta 1) faccio PCR con primers che siano per ALU, quindi prendo gli amplificati e carico su gel, faccio elettroforesi...
qui inizio a avere un problema:
faccio elettroforesi. trovo le bande: mica questo mi dice se ha amplificato?
|
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 09 ottobre 2012 : 19:40:18 Inserito il - 09 ottobre 2012 : 19:40:18




|
Citazione:
trovo le bande: mica questo mi dice se ha amplificato?
Beh, se hai una banda qualche cosa è stato amplificato, altrimenti non vedresti nulla.
Per confermare che sia la buona banda cominci a guardarne la lunghezza che deve corrispondere a quella della regione che volevi amplificare. Se poi vuoi essere sicura al 100% fai sequenziare la sequenza nella banda e ti metti l'animo in pace :D |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 09 ottobre 2012 : 22:06:34 Inserito il - 09 ottobre 2012 : 22:06:34




|
lo sai qual'è il mio dubbio? che ovvio è sbagliato, ma non capisco:
io avevo DNA, quindi, amplificato o non amplificato, io qualcosa devo vedere... poi i primers sono oligonucleotidi... qualcosa ci sta nella provetta... anche se non amplifica!
eppure io mi ricordo che in lab, quando feci la tesi, se non vedevo bande non avevo amplificato, ma non mi trovo....
comunque, mettendomi l'animo in pace, trovando le bande io ho amplificato la regione di mio interesse, che contiene ALU.
a me in quel caso specifico mi interessa solo aver trovato le sequenze ALU, per discriminare DNA umano(anche perchè tornando a quanto dice GFPina, io credo che anche se non detto, sicuramente il prof si riferisce a un ipotetico sistema dove ho DNA umano e DNA virale... alla fine è sempre così che ci ha spiegato le cose, e spesso usando l'esempio con SV40.)
quindi mi interessa solo che ha amplificato |
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
 |
|
|
Citosina
Nuovo Arrivato

Città: Milano

73 Messaggi |
 Inserito il - 09 ottobre 2012 : 23:47:25 Inserito il - 09 ottobre 2012 : 23:47:25




|
Citazione:
lo sai qual'è il mio dubbio? che ovvio è sbagliato, ma non capisco:
io avevo DNA, quindi, amplificato o non amplificato, io qualcosa devo vedere... poi i primers sono oligonucleotidi... qualcosa ci sta nella provetta... anche se non amplifica!
eppure io mi ricordo che in lab, quando feci la tesi, se non vedevo bande non avevo amplificato, ma non mi trovo....
Sul gel vedi alla fine solo l'amplificato e al massimo i dimeri dei primers che sono sempre in fondo al gel essendo di pochi bp. poco DNA non amplificato non lo vedi (o comunque non lo vedi come banda, ma al massimo come dello "smear" lungo tutta la lane..in pratica una strisciata bianca senza bande). |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 10 ottobre 2012 : 09:26:24 Inserito il - 10 ottobre 2012 : 09:26:24




|
ok... mi sono rinfrescata la memoria... infatti sapevo questa cosa dello smear!
quindi ora la mia risposta è più completa e corretta?
se vedo le bande amplificate, ho trovato le sequenze ALU?
giusto?
è sufficiente o devo completare con qualche altro passaggio?
grazie a tutti per i preziosi consigli!! |
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
 |
|
|
prossima biologa
Utente Attivo
  


1437 Messaggi |
 Inserito il - 10 ottobre 2012 : 09:28:19 Inserito il - 10 ottobre 2012 : 09:28:19




|
Citazione:
Messaggio inserito da chick80
Per la 2 io farei una banale PCR per SRY, molto più easy che discriminare 6bp...
risponderò amelogenina, se mi dovesse capitare, ma condivido e credo che accennerò alle SRY, la cui esistenza era rinchiusa in un oscuro angolo della mia memoria!!!
|
Aiuto in biochimica?guarda qui
Info sul lavoro di biologo?guarda qui |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 10 ottobre 2012 : 09:33:14 Inserito il - 10 ottobre 2012 : 09:33:14





|
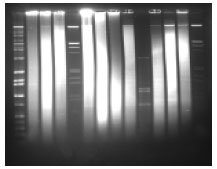
Scusate ma il DNA genomico non è affatto uno smerar, si presenta come una grossa banda ad alto peso molecolrae che migra a fatica nel gel, come si vede dall'immagine postata da chick80.
Si ha uno smear di DNA genomico in due casi:
- DNA degradato
- DNA tagliato con enzimi di restrizione, come in questa immagine:
 |
 |
|
| |
Discussione |
|








