| Autore |
Discussione |
|
|
kapyn
Nuovo Arrivato
Prov.: napoli

22 Messaggi |
 Inserito il - 23 febbraio 2007 : 17:29:30 Inserito il - 23 febbraio 2007 : 17:29:30





|
Ciao a tutti, ho l'esame di biologia molecolare la prox settimana e ho cominciato da poco con le tecniche , ci sono molte cose che nn mi sono chiare, spero che qualcuno di voi possa aiutarmi , ci sono molte cose che nn mi sono chiare, spero che qualcuno di voi possa aiutarmi
Allora,cominciamo con quale differenza c'è tra clonaggio e pcr, cioè ho capito che per la pcr devo cnosere almeno in parte la sequenza nucleotidica che voglio amplificare, giusto? Ma nn ho capito allora come si fa ad amplificare col clonaggio...cioè come faccio a selezionare l'inserto da mettere nel vettore??? nn devo cmq sapere la sequenza ? o basta sapere soltanto in quale frammento ( ottenututo dalla restrizione) si trova? AIUTOOO
|
|
|
|
|
asparagina20
Nuovo Arrivato
Prov.: Cuneo
Città: alba

28 Messaggi |
 Inserito il - 23 febbraio 2007 : 18:53:22 Inserito il - 23 febbraio 2007 : 18:53:22




|
Ciao
ache io sto preparando l'esame di bio molecolare!!
Allora, il fine della PCR è l'amplificazione di una sequenza di DNA, il clonaggio pure! Però qual è la differenza? Il mio prof ha insistito molto su questo punto, dicendo che la PCR non è una tecnica di clonaggio perchè clonare significa FORMARE UN GRUPPO DI ORGANISMI GENETICAMENTE IDENTICI FRA DI LORO. Perciò finchè non metto il gene in un vettore e infetto un organismo non posso parlare di clonaggio, ma solo di amplificazione.
Poi per quanto riguarda la scelta dell'inserto da mettere in un vettore ti posso dire ciò che ho capito io: in pratica quando io voglio mettere un inserto in un vettore, cioè fare un clonaggio, voglio avere tante copie del mio gene d'interesse. Sto gene deriva magari dal DNA di una cellula e io voglio mappare il gene responsabile di una malattia. Taglio il DNA con enzimi di restrizione e usando una sonda (SOUTHERN) cerco il mio gene. Una volta trovato tratterò con lo stesso enzima il mio vettore e inserisco il gene nel vettore, dopo di che faccio un clonaggio. La domanda più ovvia è: come costruisco la sonda??? Il fatto è che io potrei benissimo conoscere almeno parte della sequenza amminoacidica della proteina, da qui, usando il codice genetico, posso risalire a una sonda più o meno appropriata! Riassumendo: prendo DNA dalla mia cellula, faccio southern con una sonda da me creata, individuo gene, inserisco il gene nel vettore e faccio clonaggio (Cioè faccio crescere tante cellule geneticamente identiche che hanno il mio gene d'interesse). Mi sono spiegata?? Perchè ho sempre qualche problema col farmi capire!!!  CIAOOOO CIAOOOO |
paoletta |
 |
|
|
asparagina20
Nuovo Arrivato

Prov.: Cuneo
Città: alba

28 Messaggi |
 Inserito il - 23 febbraio 2007 : 18:59:32 Inserito il - 23 febbraio 2007 : 18:59:32




|
Se ho scritto qualche boiata ditemelo!!!!!!!! 
ciao |
paoletta |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 23 febbraio 2007 : 19:35:18 Inserito il - 23 febbraio 2007 : 19:35:18





|
Ciao!
Scusate ma sono un po’ di fretta cerco di fare chiarezza in due parole:
La PCR (Polymerase Chain Reaction) serve per amplificare un frammento di DNA, devo conoscere in parte la sequenza perchè devo disegnare dei primers che sono complementari a parte della sequenza che voglio amplificare. (le estremità della sequenza)
Per una spiegazione più dettagliata provate a leggere su wikipedia:
http://it.wikipedia.org/wiki/Pcr
Il clonaggio invece è fatto inserendo un frammento di DNA in un vettore, il vettore+inserto viene poi messo in Batteri (E. Coli) e fatto “amplificare” nel senso che quando il batterio si divide oltre a replicare il proprio DNA replica anche il DNA del plasmide (vettore), quindi alla fine si formano tante copie del vettore+frammento.
http://it.wikipedia.org/wiki/Clonaggio_genico
es. di vettore
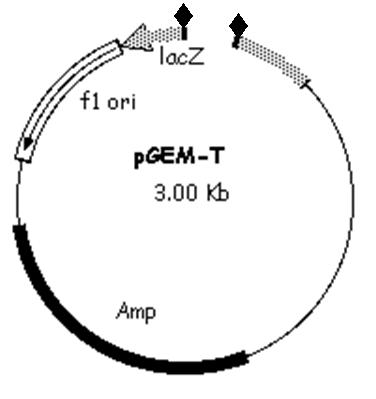
13,44 KB
Citazione:
Messaggio inserito da asparagina20
Il mio prof ha insistito molto su questo punto, dicendo che la PCR non è una tecnica di clonaggio perchè clonare significa FORMARE UN GRUPPO DI ORGANISMI GENETICAMENTE IDENTICI FRA DI LORO. Perciò finchè non metto il gene in un vettore e infetto un organismo non posso parlare di clonaggio, ma solo di amplificazione.
Perfetto!!! 
Citazione:
Messaggio inserito da asparagina20
Poi per quanto riguarda la scelta dell'inserto da mettere in un vettore ti posso dire ciò che ho capito io: ..... (SOUTHERN)...

Attenzione il Southern è un'altra storia!!! Non c'entra con il clonaggio! Non c'entra con il clonaggio!
Per clonare di sono vari metodi, quello più usato è con enzimi di restrizione: (per farla semplice, questo è un esempio)
- tagli il frammento da inserire con un enzima di restrizione (che taglia il tuo frammanto alle due estremità)
- tagli anche il vettore (circolare) con lo stesso enzima di restrizione (taglia una volta e il vettore si linearizza)
- fai una ligazione cioè fai legare il tuo frammento tagliato all'interno del vettore (che torna cirolare) e hai ottenuto vettore+frammento
Spero di aver chiarito un po', se avete dubbi chedete pure, scusate la fretta ma ora devo scappare!
Ciao |
 |
|
|
asparagina20
Nuovo Arrivato

Prov.: Cuneo
Città: alba

28 Messaggi |
 Inserito il - 23 febbraio 2007 : 19:53:27 Inserito il - 23 febbraio 2007 : 19:53:27




|
Ciao! 
Grazie mille per la risposta!! Mi sa che ho confuso lo screening della library col southern o non so che cavolo dico!! 
Il problema è: Sto cavolo di inserto da dove viene fuori?? Dallo screening di una library? Da una PCR con primer appositi? Cioè....Se io voglio cercare nel genoma un gene (ad es perchè so che è responsabile di una malattia) e poi clonarlo, devo fare una library e vedere dov'è il gene. Dopo di che lo metto in un vettore per clonarlo, giusto? |
paoletta |
 |
|
|
kapyn
Nuovo Arrivato
Prov.: napoli

22 Messaggi |
 Inserito il - 23 febbraio 2007 : 20:26:55 Inserito il - 23 febbraio 2007 : 20:26:55





|
Aspettate, penso di essere ancora + confusa di prima.
Quello che nn mi è chiaro, è l'INSERTO!
cioè, qua mi dice che posso fare il clonaggio prendendo un frammento, oppure usando tutto il genoma frammentato per fare la libreria...consideriamo il primo caso:
voglio isolare un gene...COME FACCIO??? cioè se non conosco la sequenza(altrimenti facevo la PCR), come faccio a risalire a quel determinato gene? Anch'io avevo pensato che vi si risaliva conoscendo per esempio la proteina o dall'mRNA , costruisci le sonde e fai l'ibridazione coi frammenti magari ottenuti dal trattamento con un enzima di restrizione...o no? , costruisci le sonde e fai l'ibridazione coi frammenti magari ottenuti dal trattamento con un enzima di restrizione...o no?
Se faccio la library il problema è lo stesso, cioè come si fa lo screening se nn so com'è fatto il gene? anche qui mi viene in mente la sonda come unica soluzione,,,bah nn lo so...  |
 |
|
|
asparagina20
Nuovo Arrivato

Prov.: Cuneo
Città: alba

28 Messaggi |
 Inserito il - 23 febbraio 2007 : 20:53:26 Inserito il - 23 febbraio 2007 : 20:53:26




|
Appunto, se conosco la proteina faccio una sonda degenerata (perchè alcuni amminoacidi sono determinati da più codoni) e faccio un'ibridazione ad alta stringenza, così ricavo il gene.
Per esempio: ho cristallizzato la proteina, so la sequenza, vado a cercare un pezzo della sequenza in cui ci sono un bel po' di amminoacidi che sono determinati da 1, massimo 2 codoni, faccio tutte le sonde possibili (così ovvio il problema della degenerazione del codice) e le uso per ibridare ad alta stringenza una library!! Trovo il gene e lo clono!! Secondo me è così...in classe ci avevano fatto lo stesso discorso per il fattore VIII come esempio di individuazione di un gene! Mah!!  |
paoletta |
 |
|
|
Balsamo del Canadà
Nuovo Arrivato

Prov.: Verbano-Cusio-Ossola
Città: Verbania

97 Messaggi |
 Inserito il - 24 febbraio 2007 : 10:48:07 Inserito il - 24 febbraio 2007 : 10:48:07




|
buongiorno a parte che spero di non essere l'unico idiota che il sabato è a casa a studiare volevo dirvi che per trovare un gene da una libreria ci sono metodi differenti. Quando è possibile avere una parte di sequenza allora non ci dovrebbero essere problemi ma non è proprio un southern che si usa, è una ibridazione su filtro. Praticamente si fanno aderire le colonie ad un filtro, si rompono le cellule in condizioni denaturanti e poi ci appiccico la sonda in condizioni più o meno stringenti(a seconda della sonda). Con l'autoradiografica o meglio adesso si usano sonde fluorescenti vaado a cercare la colonia che da il segnale per poi recuperarla facilmente dalle piastre madri.
Però voglio ricordare che seppur in maniera più difficile è possibile recuperare il frammento con un gene specifico anche non sapendone la sequenza. Un esempio lampante sono tutti gli studi genetici basati sulla complementazione. Quando un ceppo mutato in una certa funzione (per esempio gli è stato deleto un gene, oppure ha una mutazione termosensibile e cioè che si manifesta solo a determinate temperature) viene trasformato con una libreria e poi piastrato in condizioni selettive avremo che solo il vettore con il frammento che ripristina la funzione mutata permetterà la crescita della colonia.
Questa è una tecnica largamente usata ma dico che è un pò più complicata perchè è necessario che le sequenze regolative siano intatte e soprattutto riconosciute nell'organisco in cui stiamo lavorando.
Spero di essere stato di aiuto. |
-B.del C.- |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 24 febbraio 2007 : 11:14:19 Inserito il - 24 febbraio 2007 : 11:14:19





|
Ciao ragazze,
allora una volta stabilito cos’è il clonaggio (che spero sia chiaro), come ho accennato velocemente ieri ci sono varie strategie di Clonaggio.
Il “frammento” di cui parlavo può essere ottenuto in diversi modi:
ad es.
- si taglia il DNA con enzimi di restrizione, ottengo tanti frammenti e posso inserire ogni frammento in un vettore … colaggio… ottengo una libreria
- conoscendo la sequenza posso amplificare il frammento che mi interessa mediante PCR… ho il mio frammento da inserire nel vettore… clonaggio
Ecc…
Poi come dice giustamente asparagina20 posso avere il caso in cui conosco la sequenza e quello in cui non la conosco… e anche qua posso conoscere la sequenza proteica ma non quella aminoacidica… a questo punto è giusto quello che avete detto sulle sonde. Qua c’è il discorso dello screening delle librerie.
Siccome il discorso diventa un po’ complesso vi consiglio di dare un’occhiata a questi link, che vi spiegano le cose molto meglio di quanto non possa fare io:
1) semplice descrizione della tecnologia del DNA ricombinante (per avere un’idea iniziale!)
http://scuole.provincia.ps.it/ls.laurana/settimana/tecnologia_del_dna_ricombinante.htm
2) sito dove sono presenti la lezioni (ppt) di “Biotecnologie Ricombinanti” della Università La Sapienza di Roma (personalmente le trovo fatte molto bene ed esplicative)
http://w3.uniroma1.it/biotecric/lezioni.htm
Vi consiglio in particolare queste lezioni:
Introduzione alle Biotecnologie Ricombinanti: http://w3.uniroma1.it/biotecric/lez_NN1.ppt
Vettori di clonaggio: http://w3.uniroma1.it/biotecric/lez_NN5.ppt
Selezione e analisi di cloni ricombinanti: http://w3.uniroma1.it/biotecric/lez_NN6.ppt
Librerie geniche in vitro: http://w3.uniroma1.it/biotecric/lez_NN7.ppt
Strategie di clonaggiohttp://w3.uniroma1.it/biotecric/lez_NN8.ppt
Spero siano utili per chiarire tutti i dubbi
Buono Studio
 |
 |
|
|
asparagina20
Nuovo Arrivato

Prov.: Cuneo
Città: alba

28 Messaggi |
 Inserito il - 24 febbraio 2007 : 11:56:26 Inserito il - 24 febbraio 2007 : 11:56:26




|
Grazie mille a tutti!!! :) Siete stati moooolto gentili!! 
Spero solo che mi vada bene sto cavolo di esame!! CIAOOO buon week end |
paoletta |
 |
|
|
kapyn
Nuovo Arrivato
Prov.: napoli

22 Messaggi |
 Inserito il - 26 febbraio 2007 : 09:08:55 Inserito il - 26 febbraio 2007 : 09:08:55





|
grazie! finalmente ho capito qlcosina in +! |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 26 febbraio 2007 : 10:01:22 Inserito il - 26 febbraio 2007 : 10:01:22





|
Beh a questo punto:
in bocca al lupo ad entrambe per l'esame!
Fateci sapere.
Ciao  |
 |
|
|
kapyn
Nuovo Arrivato
Prov.: napoli

22 Messaggi |
 Inserito il - 01 marzo 2007 : 12:30:48 Inserito il - 01 marzo 2007 : 12:30:48





|
 E'ANDATA!!!27!!! E'ANDATA!!!27!!!  
Pensavo di nn farcela, invece!SONO TROPPO CONTENTA!
Grazie ancora, questo forum mi è stato molto d'aiuto!
|
 |
|
|
asparagina20
Nuovo Arrivato

Prov.: Cuneo
Città: alba

28 Messaggi |
 Inserito il - 01 marzo 2007 : 19:59:13 Inserito il - 01 marzo 2007 : 19:59:13




|
Anche a me è andata!! Ho preso 30!!!   
Sono strafelice!! Grazie a tutti!!  
Ciaoooooooooooo |
paoletta |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 02 marzo 2007 : 16:37:16 Inserito il - 02 marzo 2007 : 16:37:16





|
Complimenti ad entrambe!!!!
            |
 |
|
| |
Discussione |
|





 CIAOOOO
CIAOOOO

 , costruisci le sonde e fai l'ibridazione coi frammenti magari ottenuti dal trattamento con un enzima di restrizione...o no?
, costruisci le sonde e fai l'ibridazione coi frammenti magari ottenuti dal trattamento con un enzima di restrizione...o no? E'ANDATA!!!27!!!
E'ANDATA!!!27!!!