| Autore |
Discussione |
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 18 giugno 2008 : 19:46:48 Inserito il - 18 giugno 2008 : 19:46:48




|
ciao a tutti, ho un pò di problemi con il sequenziamento e ogni volta che chiedo a voi poi ho non solo buoni consigli ma anche fortuna quindi....chiedo!
vorrei sapere se la dimensione del frammento che io posso sequenziare dipende dalla lunghezza del capillare che ho io nel sequenziatore che uso? il mio è lungo 36 cm, qualcuno sa dirmi quanto possono essere lunghi al massimo i miei amplificati? credo di avere degli amplificati troppo lunghi perchè li vedo bene fino a 400-500 bp ma poi sono un pò brutti da analizzare e con l'R non riesco a coprire tutto il tratto di interesse quindi non so benen che fare se non ridisegnarmi i primer e fare amplificati più piccoli....
grazie mille ciao
ps avete qualche sito carino da consigliarmi per imparare un pò di cose sul sequenziamento?
graaaazie ciaoooo
|
|
|
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 18 giugno 2008 : 20:03:44 Inserito il - 18 giugno 2008 : 20:03:44




|
forse avreidovuto specificare che ho in dotazione in lab un sequenziatore automatico ABI PRISM 3100 a 16 capillari.....e uso il polimero POP4....per me è scontato ma mi rendo conto che sono un pò imprecisa
ciao |
 |
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 19 giugno 2008 : 21:48:57 Inserito il - 19 giugno 2008 : 21:48:57




|
ma proprio nessuno mi risponde??? |
 |
|
|
rosita84
Nuovo Arrivato

11 Messaggi |
 Inserito il - 20 giugno 2008 : 01:43:41 Inserito il - 20 giugno 2008 : 01:43:41




|
| ciao (premetto ke nn ho mai sequenziato) ma secondo me non e' la lunghezza della colonna il tuo problema.....la lunghezza dei frammenti sequenziati non puo' essere mai maggiore di 500bp e quindi in quel caso dovrai disegnare piu' coppie di primer x riuscire a ricostruire (mettendo insieme) le seuqnze ottenute ed arrivare cosi' anke a 1000 bp!!!Non so se sono stata chiara |
 |
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 22 giugno 2008 : 00:31:11 Inserito il - 22 giugno 2008 : 00:31:11




|
ciao, grazie sei stata chiarissima. non sono d'accordo però....la lunghezza dei frammenti può abbondantemente superare ile 500bp, non so se io non riesco a leggerle a causa della lunghezza del mio array e del polimero che uso...o boh 
ciaoo |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 22 giugno 2008 : 01:35:24 Inserito il - 22 giugno 2008 : 01:35:24





|
e' il sequenziatore che "sfarfalla" dopo le 500 bp,dovresti usare primer interni
non ti so dire di piu' su come si usa perche' da noi va di moda che chi sa le cose se le tiene per se' .... |

|
 |
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 23 giugno 2008 : 19:20:03 Inserito il - 23 giugno 2008 : 19:20:03




|
ciao Patrizio, grazie per le informazioni, allora non sono io che faccio pasticci  ma è il mitico sequenziatore!!! ma è il mitico sequenziatore!!!
però non so che cosa intendi quando mi dici di usare primer interni, forse dalle seq che ottengo mi devo disegnare altri primer e così via? mi sa che dovrò procedere così peccato però....
che brutto quando le persone non condividono, sono insicure evidentemente!! purtroppo ovunque è un pò così....
 |
 |
|
|
rosita84
Nuovo Arrivato

11 Messaggi |
 Inserito il - 24 giugno 2008 : 02:11:33 Inserito il - 24 giugno 2008 : 02:11:33




|
| allora fai come vuoi.....in bocca al lupo |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 24 giugno 2008 : 08:46:45 Inserito il - 24 giugno 2008 : 08:46:45





|
Citazione:
Messaggio inserito da rosita84
la lunghezza dei frammenti sequenziati non puo' essere mai maggiore di 500bp e quindi in quel caso dovrai disegnare piu' coppie di primer x riuscire a ricostruire (mettendo insieme) le seuqnze ottenute ed arrivare cosi' anke a 1000 bp!!!Non so se sono stata chiara
a 1000 bp ci arrivi gia' con 2 primer  500 + 500 , 500 + 500 ,
Citazione:
Messaggio inserito da ameliap
però non so che cosa intendi quando mi dici di usare primer interni, forse dalle seq che ottengo mi devo disegnare altri primer e così via?
si amelia intendevo questo,tu ottieni la prima sequenza del tuo clone...poi dovresti ordinare altri primer e overlappi
|

|
 |
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 25 giugno 2008 : 22:56:21 Inserito il - 25 giugno 2008 : 22:56:21




|
grazie Patrizio, farò così però ci metterò molto più tempo,tra disegnare i primer ( ci metto poco uso primer 3..)chiedere ok per comperarli e comperarli.... peccato ma se è necessario fare così....
 |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 25 giugno 2008 : 23:36:17 Inserito il - 25 giugno 2008 : 23:36:17





|
questa e' una delle mie sequenze..come vedi anche fino a 670 si legge abbastanza bene,un altra cosa che mi viene in mente( io non l ho mai fatto ) e' fare magari piu' clonaggi usando sempre gli stessi primer del vettore..se stai usando quello..ovviamente se il tuo inserto e' 2 kb e hai la fortuna di trovare un sito di restrizine nella sequenza e' fatta  ..cosi' non "scocci" con gli ordini ..cosi' non "scocci" con gli ordini
Immagine:
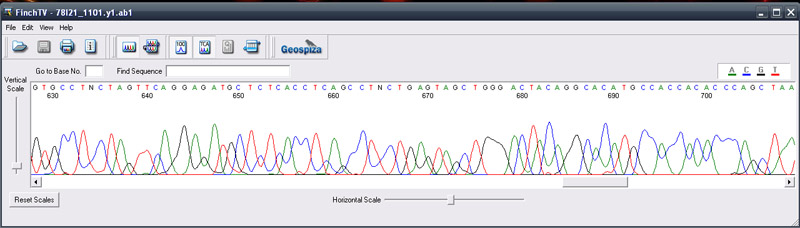
88,93 KB |

|
 |
|
|
ameliap
Nuovo Arrivato

32 Messaggi |
 Inserito il - 26 giugno 2008 : 20:20:06 Inserito il - 26 giugno 2008 : 20:20:06




|
ciao Patrizio,
la tua sequenza è davvero molto bella, le mie non lo sono mai così fino a 600....com'è il tuo protocollo della reazione di sequenza nel termociclatore? anche tu usi un 3100 con il POP4? anche tu usi un 3100 con il POP4?
 |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 26 giugno 2008 : 22:25:06 Inserito il - 26 giugno 2008 : 22:25:06





|
ameliap in questo periodo non sono in lab perche' mi sono preso del tempo per fare un esame e sinceramente non mi ricordo ne il tipo di sequenziatore ne il resto perche' ho il mio laboratory book li  |

|
 |
|
|
Topogigio77
Nuovo Arrivato

Città: Milano

37 Messaggi |
 Inserito il - 04 luglio 2008 : 12:45:29 Inserito il - 04 luglio 2008 : 12:45:29




|
Ciao ameliap,
scusa se entro solo ora nella discussione...ecco come la vedo io:
- puoi tranquillamente arrivare a 600-650 bp ma con Capillary Array nuovi, POP4 fresco e strumento ben tenuto (lavaggi settimanali e buffer cambiato ogni 2 giorni, laser non vecchio come il cucco)
- per aumentare la lettura basta cambiare la lunghezza dei capillari: con quello da 50cm arrivi a 800-850 basi. L'unica cosa è che devi fare la matrice (calibrazione spettrale) se non è stato calibrato sui 50cm. Se hai dubbi fammi sapere
- se vuoi arrivare a 1000bp senza rifare i primer ci sono i capilalri da 80cm...buon divertimento... ;o)
In questo caso consiglio anch'io di spezzare in 2 l'amplicone...
spero possa servire. ciao |
 |
|
|
E. Coli
Nuovo Arrivato


22 Messaggi |
 Inserito il - 04 luglio 2008 : 15:25:15 Inserito il - 04 luglio 2008 : 15:25:15




|
| scusate se mi intrometto anche se ho poca esperienza ma la lunghezza di DNA sequenziato può anche arrivare a più di 1000bp. io ho costruito dei mutanti di delezione e con un primer ho sequenziato 1235 bp. il punto sta nell'avere un DNA purificato che non presenti tracce dei buffers e delle colonnine usate per la purificazione. inoltre se il DNA lo purifichi in seguito a inoculo di una colonia pescata da una piastra petri di LB solido è assolutamente essenziale che tu sia certa di pescare SOLO quella colonia. Spesso, se non osservi attentamente la petri, intorno alla tua colonia ci sono piccolissime colonie satellite che vanno ad inficiare con la presenza del loro DNA nel purificato, la lunghezza e la bontà della tua sequenza. |
 |
|
| |
Discussione |
|








