Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
ElisabettaCardiff
Nuovo Arrivato

6 Messaggi |
 Inserito il - 18 maggio 2009 : 15:49:06 Inserito il - 18 maggio 2009 : 15:49:06




|
Ciao a tutti!
Allora, domandina...
Dobbiamo fare un qPCR. E` la prima volta che lo usiamo e qui non c`e` nessuno che l`abbia usato.
Quindi abbiamo il manuale di istruzioni, qualche articolo e...tanta buona volonta`!
Dobbiamo, prima di procedere fare una curva di standardizzazione? In ascissa la concentrazione di DNA (ovviamente diverse diluizioni) e in ordinata il numero di cicli che la macchina utilizza???
E qui nasce un problemino... Per valutare la concentrazione del DNA c`e` qualcosa all`interno della macchina? O basta affidarsi a un normalissimo spettrofotometro con programma di riconoscimento DNA???
GRAZIE!!!!!!!!
Betta
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 18 maggio 2009 : 23:39:07 Inserito il - 18 maggio 2009 : 23:39:07





|
Ciao Elisabetta,
mi sembra che sia un po' di confusione sull'utilizzo della qPCR.
Cerco di fare un po' di chiarezza.
Allora il tutto dipende a cosa vi serve la qPCR.
Cosa dovete quantificare DNA o cDNA?
Che tipo di quantificazione dovete fare? Assoluta o relativa?
Intendo:
Quantificazione assoluta: si utilizza una curva standard con concentrazioni di DNA noto, in questo modo è possibile quantificare i campioni in modo assoluto estrapolando i valori di concentrazione dalla curva standard. Questo è utilizzato ad es. quando si vogliono valutare le copie di DNA virale presente nei campioni.
Per studi di espressione genica generalmente si utilizza la quantificazione relativa.
Quantificazione relativa: si utilizza un gene housekeeping (HK) per normalizzare i valori. Presupponendo che il gene housekeeping sia espresso allo stesso livello in tutti i campioni. Normalizzando rispetto all’housekeeping vedi se il tuo gene (GOI) è espresso a livello maggiore ad esempio in campioni trattati rispetto ai controlli.
Questo però sottintende un presupposto: che l'efficienza delle delle 2 reazioni di PCR, quella dell'housekeeping e quella del tuo gene, sia uguale. Ovviamente non è così. Però quello che viene fatto è ogni volta che si utilizza una coppia di primers è testarne l'efficienza. Questo viene fatto utilizzando una curva standard in questo modo:
- prendi un campione in cui siano espressi sia l'HK che il GOI
- fai delle diluizioni seriali di questo campione
- fai una real-time costruendoti una curva standard di queste diluizioni (ovviamente la curva ti darà dei valori "relativi" non "assoluti"!)
- valuti l'efficienza delle due PCR che deve essere attorno a 1 (slope attorno 3,3) e deve essere molto simile per le 2 PCR
Fatto questo puoi procedere ad analizzare i tuoi campioni e quantificarli mediante quantificazione relativa.
(la curva standard in questo caso va fatta solo una volta per valutare l'efficienza, non devi ripeterla poi nelle analisi successive)
Il metodo che si utilizza per fare questa quantificazione in genere è quello del deltadeltaCt.
prima calcoli il deltaCt conftontando il tuo gene con l'housekeeping (nello stesso campione):
deltaCt = Ct GOI - Ct HK
poi calcoli il deltadeltaCt, in questo caso hai bisogno di un campione di riferimento (ad es. poniamo campione non trattato) che poini uguale a 1 e valuti un incremento o decremento dell'espressione del tuo gene nei tuoi campioni rispetto al controllo (campione di riferimento)
deltadeltaCt= deltaCt Sample - deltaCt Reference
la variazione sarà data da 2^(-deltadeltaCt)
Detto questo per rispondere comunque ai tuoi dubbi:
Citazione:
In ascissa la concentrazione di DNA (ovviamente diverse diluizioni) e in ordinata il numero di cicli che la macchina utilizza???
si più o meno è così, in ascissa hai "la concentrazione di DNA", utilizzi un DNA a concentrazione nota oppure "il logaritmo della quantità iniziale"
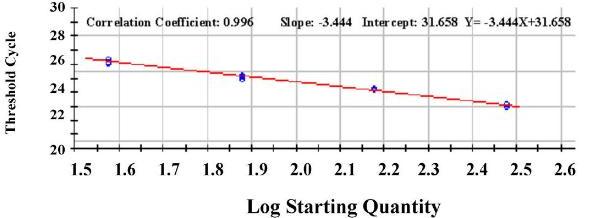
Citazione:
Per valutare la concentrazione del DNA c`e` qualcosa all`interno della macchina? O basta affidarsi a un normalissimo spettrofotometro con programma di riconoscimento DNA???
No all'interno della macchina non c'è niente!
Le opzioni sono:
- fai una quantificazione assoluta partendo da quantità note di DNA e in questo caso o compri degli standard a concentrazioni note, oppure puoi quantificare il tuo DNA utilizzando altri metodi di quantificazione, nota che l'unico veramente affidabile è quello "fluorimetrico", quello "spettrometrico" non è sufficientemente affidabile!
- fai la curva standard partendo da diluizioni del tuo cDNA, in questo caso assegni tu dei valori di concentrazione ad es.
cDNA indiluito 1
cDNA 1:2 0,5
cDNA 1:4 0,25...
infine ti rimando ad alcune discussioni sul forum, in cui si è gia parlato di questo argomento:
http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=7440
http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=1423
http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=3813
e questo link dove è spiegato tutto più in dettaglio ed è spiegato anche come si arriva alla formula: 2^(-deltadeltaCt)
http://www.uic.edu/depts/rrc/cgf/realtime/data.html
spero sia un po' più chiaro, se hai altri dubbi scrivi pure!
  |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|





