| Autore |
Discussione |
|
|
jeCO85
Nuovo Arrivato

51 Messaggi |
 Inserito il - 17 gennaio 2009 : 14:43:36 Inserito il - 17 gennaio 2009 : 14:43:36




|
Scusate la domanda magari molto ignorante e banale ma mi è venuto un dubbio:
se mando a sequenziare in service un amplificato che ritengo possa avere una delezione di poche basi in eterozigosi (io ho l'amplificato indiscriminato di entrambi gli alleli) che tipo di risultato mi viene fornito?
|
|
|
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:12:28 Inserito il - 17 gennaio 2009 : 16:12:28





|
| non ti appare la delezione |

|
 |
|
|
jeCO85
Nuovo Arrivato

51 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:29:41 Inserito il - 17 gennaio 2009 : 16:29:41




|
E' infatti purtroppo quello che immaginavo  (però non intendendomi di sequenziamento pensavo che magari esisteva un metodo per valutare la quantità di basi sequenziate e da cui discriminare quindi se si tratta di due amplificati analoghi o diversi). (però non intendendomi di sequenziamento pensavo che magari esisteva un metodo per valutare la quantità di basi sequenziate e da cui discriminare quindi se si tratta di due amplificati analoghi o diversi).
Credo comunque che dovrò sequenziare per vedere se ci sono polimorfismi o mutazioni in sequenza.
Nel caso comunque vi fosse veramente la delezione di poche basi ritenete sia possibile vederla usando la SSCP? Tenete conto che non ho riferimenti "patologici" e "sani", mi interessa solo vedere se c'è differente motilità (e poi magari prendere le bande diverse e sequenziare quelle).
Però per la SSCP cosa mi serve? Con cosa marco il DNA a singola elica? E soprattutto, è possibile farla con un amplificato sulle 400-500 bp?
Grazie ancora! |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:39:43 Inserito il - 17 gennaio 2009 : 16:39:43





|
Purtroppo è così. La delezione non appare perchè quello "spazio vuoto" lasciato
dall'amplicone deleto viene riempito, nella corsa capillare, dall'altro amplicone.
Al limite potresti vedere un dimezzamento dell'altezza dei picchi presso quelle
due/tre (?) basi che supponi possano essere delete. Però non te l'assicuro,
l'elettroferogramma non è tecnica quantitativa e potrebbero intervenire altre
variabili a determinare l'area alla base dei picchi.
Forse un gel di poliacrilamide potrebbe essere la soluzione migliore,
ma dipende da qual'è l'entità della delezione (più grande è, meglio risolvi).
Sulla SSCP purtroppo non so dirti nulla. |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
jeCO85
Nuovo Arrivato

51 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:42:48 Inserito il - 17 gennaio 2009 : 16:42:48




|
Hai ragione, non avevo pensato all'alternativa del gel di acrilamide!
con quello, se c'è la delezione anche di 2-3 nucleotidi, dovrei già vederla osservando due bande diverse.
Grazie del suggerimento :) |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:50:17 Inserito il - 17 gennaio 2009 : 16:50:17





|
per quanto riguarda il gel se sono 2-3 nt ,preparati a farne uno bello, forse anche all 8% ,a fare una corsa leeeentissima tipo 50 V e di aspettare anche piu' di mezza giornata
Io non farei molto affidamento ai picchi in questo caso... |

|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:55:57 Inserito il - 17 gennaio 2009 : 16:55:57





|
| eheh più che altro preparati a rifarlo taaaaaaante volte :P |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 17 gennaio 2009 : 16:58:18 Inserito il - 17 gennaio 2009 : 16:58:18





|
 sadico sadico |

|
 |
|
|
jeCO85
Nuovo Arrivato

51 Messaggi |
 Inserito il - 17 gennaio 2009 : 17:02:31 Inserito il - 17 gennaio 2009 : 17:02:31




|
| speriamo piuttosto che ci sia veramente qualcosa (è una storia moooolto lunga...)! e che sia una sostituzione di basi così la vedo in sequenza! |
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 00:25:16 Inserito il - 18 gennaio 2009 : 00:25:16




|
guarda che la delezione si vede eccome...ti appare la sequenza dell allele deleto in sovrapposizione allallele non deleto a partire dal punto in cui inizia la delezione...se invece la sequenza legge due wt o cmq qualcosa in omozigosi allora avrai perfetta sovrapposizione dei picchi e ti tocca confrontare la tua sequenza con una wt... esiste un programma che si chiama seqman che allinea la sequenz di rifermento che insersci tu con quelle dei campioni
altrimenti che senso avrebbe sequenziare se non si vedono nemmeno le delezioni????
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 18 gennaio 2009 : 01:33:44 Inserito il - 18 gennaio 2009 : 01:33:44





|
Credo di aver detto una cavolata sulle delezioni.
Non vedresti alcun dimezzamento "locale" dei picchi, perchè la
sequenza deleta correrebbe in anticipo di 2-3 basi rispetto
a quella wt. Piuttosto avresti da un momento con l'altro
una divergenza delle due sequenze, con sovrapposizione di
basi diverse (sfasate di 2-3 nt) ed un dimezzanti di ciascuno
dei due segnali divergenti, a partire dal punto della delezione.
Almeno in teoria: nella realtà non so il risultato sarà così chiaro .
Solo con un PAGE puoi veramente risolvere due amplicono di peso diverso. |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 18 gennaio 2009 : 01:57:53 Inserito il - 18 gennaio 2009 : 01:57:53





|
mo-lab
Quindi con seqman riesci a "traslare" la sequenza
anticipata (deleta) e a sovrapportla con la wt? |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 18 gennaio 2009 : 09:27:03 Inserito il - 18 gennaio 2009 : 09:27:03





|
Citazione:
Messaggio inserito da mo-lab
guarda che la delezione si vede eccome...ti appare la sequenza dell allele deleto in sovrapposizione allallele non deleto a partire dal punto in cui inizia la delezione...se invece la sequenza legge due wt o cmq qualcosa in omozigosi allora avrai perfetta sovrapposizione dei picchi e ti tocca confrontare la tua sequenza con una wt... esiste un programma che si chiama seqman che allinea la sequenz di rifermento che insersci tu con quelle dei campioni
altrimenti che senso avrebbe sequenziare se non si vedono nemmeno le delezioni????
Si come no...
mo-lab ma seqman e' solo un programma di allineamnento !
che sequenzatore avete voi ?
i sequenziatori danno un unica sequenza..e basta,poi del file che ti esce puoi farne cio' che vuoi,
con che principio il sequenziatore dovrebbe discriminarti i 2 frammenti?
Lo strumento a parer mio ...ti darebbe una N gigante in quanto non saprebbe li in quella posizione che base ci sta
Le delezioni le vedi..ma tu usi seqman ,prendendo dalla banca dati la sequenza come riferimneto
poi 2-3 nt ma scherziamo?? no no ...non vedi proprio una mazza ,e nemmento mi fiderei ....
fatti un bel gel di agarosio o acrilammide... e spotta la banda,e sei appostissima
Da una rapida ricerca sulla rete :
Le sequenze ottenute mediante sequenziamento vengono controllate grazie al programma CHROMAS
2.13 (Technelysium Pty. Ltd.) che permette di visualizzare l’elettroferogramma delle sequenze stesse e
dunque controllare se esse sono pulite (cioè prive di background) e hanno una buona risoluzione dei
picchi.
Dopo aver controllato le sequenze, queste vengono allineate mediante i programmi SEQMAN |

|
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 13:20:53 Inserito il - 18 gennaio 2009 : 13:20:53




|
so cosa sono seq man e chromas e a cosa servono...pero io dico questo se ho la pcr di un esone ad esempio...e la metto nel sequenziatore assieme al primer f o r ..se nn ho mutazioni mi da un unica sequenza....se ho una delezione in uno degli alleli ho sovrapposizione da quel punto in poi...ad esempio ho la seq
agctgatcga e la delezione di tcg su un degli alleli
il sequenziatore (3130xl) LEGGERA:
agctgaTGCaaaatttt
agctgaaaattt....e quindi hai sovrapposizione....
come dice dyonisos...
poi allineando la sequenza tua che puoi vedere sia con chromas che con seqman con una wt presa dalle banche dati puoi vedere DOVE hai la delezione ..seqman ti aiuta ad a trovare il punto preciso perchè comedice patrizio di alliena le sequenze
|
 |
|
|
jeCO85
Nuovo Arrivato

51 Messaggi |
 Inserito il - 18 gennaio 2009 : 13:25:39 Inserito il - 18 gennaio 2009 : 13:25:39




|
Quindi secondo te mo-lab sarebbe possibile anche con pochi nucleotidi; ma il discorso che fai è valido per qualsiasi tipo di sequenziatore automatico o parli per il tuo modello?
Comunque per tagliare la testa al toro penso di mandare una mail a chi mi farà la sequenza (la Primm) e chiedergli se possono fare una cosa del genere |
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 13:27:35 Inserito il - 18 gennaio 2009 : 13:27:35




|
| certo è possibile con qualunque sequenziatore se no scusami che genere di mutazioni si potrebbero vedere cosi?solo le puntiformi?io uso sequenz per trovare ogni genere di mutazione .. |
 |
|
|
jeCO85
Nuovo Arrivato

51 Messaggi |
 Inserito il - 18 gennaio 2009 : 13:32:07 Inserito il - 18 gennaio 2009 : 13:32:07




|
ok grazie del parere mo-lab e scusami per l'ignoranza hehe ma non ho mai usato un sequenziatore, ne conosco i principi ma su questo aspetto della delezione in eterozigosi non ero sicuro della possibilità.
Grazie e buon lavoro!  |
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 13:36:30 Inserito il - 18 gennaio 2009 : 13:36:30




|
la delezione in eterozigosi la vedi ancora meglio dando un occhiata rapida all elettroferogramma anche solo con chromas perchè hai sovrapposizione dei due alleli e quindi da un certo punto in poi avrai picchi sovrapposti ...invece per le omozig hai bisogno di controllare con una sequenza di riferimento xkè gli alleli sono uguali...cmq io uso3tipi di sequenziatori diversi e funzionano tutti allo stesso modo.
ciao in bocca al lupo |
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 13:41:17 Inserito il - 18 gennaio 2009 : 13:41:17




|
PER RISPONDERE A PATRIZIO
i sequenziatori danno un unica sequenza..e basta,poi del file che ti esce puoi farne cio' che vuoi
con che principio il sequenziatore dovrebbe discriminarti i 2 frammenti?
CON IL PRINCIPIO CHE SE HAI UNA DELEZIONE ANCHE DI UNA SOLA BASE LE SEQUENZE NON SONO PIU ALLINEATE PERFETTAMENTE E AVRAI LO SFALSAMENTO DI UNA SULL ALTRA
Lo strumento a parer mio ...ti darebbe una N gigante in quanto non saprebbe li in quella posizione che base ci sta
LO STRUMENTO NON DA NESSUNA N GIGANTE MA LA NOMENCLATURA (K,M,S,R...)DATA DALLA SOVRAPPOSIZIONE DELLE BASI (AD ESEMPIO G PIU C DA ad esempio K)
Le delezioni le vedi..ma tu usi seqman ,prendendo dalla banca dati la sequenza come riferimneto
OVVIO SE NO COME FACCIO A SAPERE DOVE CADE LA MUTAZIONE?
poi 2-3 nt ma scherziamo??
COME DICEVO PRIMA NE VEDE ANCHE UNA SOLA DI BASE...
CIAO |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 18 gennaio 2009 : 14:10:01 Inserito il - 18 gennaio 2009 : 14:10:01





|
Se hai già visto elettroferogrammi di delezioni in eterozigosi
posso domandarti, molab, se si riesce a notare il dimezzamento
dei picchi ? (dal punto della delezione in poi, ovviamente)
Oppure i picchi K, M , S e R saranno ottenuti come somma? |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 18 gennaio 2009 : 14:11:36 Inserito il - 18 gennaio 2009 : 14:11:36





|
| E soprattutto: il risultato è sempre chiaro e univoco? |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 18 gennaio 2009 : 14:49:28 Inserito il - 18 gennaio 2009 : 14:49:28





|
Allora per fare questo discorso innanzitutto devi preparare la marcatura con un F e R in 2 reazioni ovviamente diverse,quindi mandi non una sequenza ma sono 2 sequenze... dal punto in cui hai la delezione in poi hai lo shift delle 2 sequenze quindi avrai una situazione simile
Immagine:
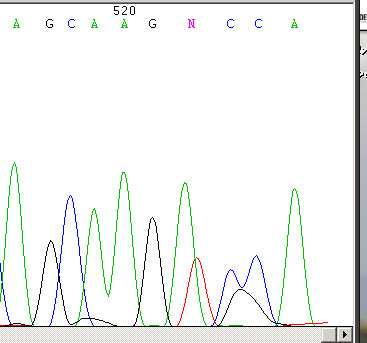
66,92 KB
poi vedi dove e' iniziato lo shft da una parte e dall altra e puoi capire dove c'e' eventualmente la mutazione
augurandoti che la mutazione si trovi pressocche' al centro del tuo frammento
|

|
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 14:50:29 Inserito il - 18 gennaio 2009 : 14:50:29




|
il risultato chiaro e univoco ce lhai mandando anche l altro strand (cioè mandando sia fow che rev)
l altezza dei picchi dipende invece dalla quantita dell allele mutato rispetto al wt e viceversa...cmq r k s sono dati dalla somma del segnale dei due picchi
quind patrizio nn stai dicendo quello che dico io?sequenziando la vedi la delezione....... |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 18 gennaio 2009 : 14:54:01 Inserito il - 18 gennaio 2009 : 14:54:01





|
e comunque e' dispendioso per chi manda le sequenze,quindi io ritengo che per una prima analisi piu' rapida ed economica il gel
|

|
 |
|
|
mo-lab
Utente Junior

Città: utrecht

348 Messaggi |
 Inserito il - 18 gennaio 2009 : 14:57:24 Inserito il - 18 gennaio 2009 : 14:57:24




|
allora prima di trattare me come se dicessi fesserie informati TU (patrizio)
mandare due sequenze costa 20euro fare un gel al3% 4ore...e nn saprai mai dov'è il punto preciso della delezione
se invece hai idea di che mutazione sia ti consiglio magari una digestione se si crea o meno un sito per qualche enzima
ciao |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 18 gennaio 2009 : 20:45:26 Inserito il - 18 gennaio 2009 : 20:45:26





|
mol-lab stai calmina,non ho motivo di trattarti come una che dice fesserie ..
ma stiamo qui per esprimere i nostri pareri ,io ragionandoci sono arrivato alle mie conclusioni,discordanti dalle tue ..pero' non te la prendere su'
 |

|
 |
|
|
Discussione |
|







