| Autore |
Discussione |
|
|
Fricerca
Nuovo Arrivato

3 Messaggi |
 Inserito il - 24 luglio 2018 : 17:03:19 Inserito il - 24 luglio 2018 : 17:03:19



|
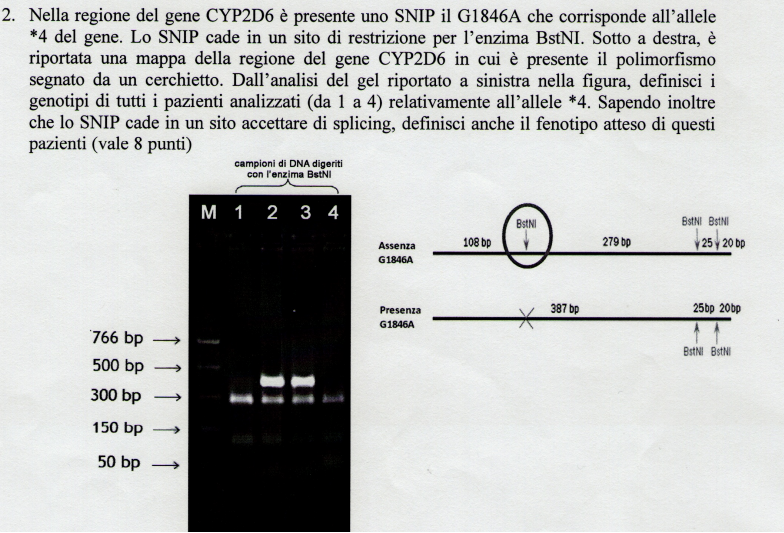
Ciao a tutti.
Tra pochi giorni ho l'esame di genetica molecolare e trovo difficoltà a risolvere un esercizio, che allego.
Secondo la mia risoluzione:
1 e 4: omozigoti per l'assenza dello SNP
2 e 3: eterozigoti
Tuttavia le bande non sono così chiaramente visibili, per cui ho dei dubbi.
Inoltre chiede il fenotipo nel caso in cui lo SNP cada in un introne.
Io credo che gli omozigoti che non hanno lo SNP saranno sani, mentre gli eterozigoti potranno avere problemi dal momento che se lo SNP cade in un sito accettore di splicing, lo splicing potrebbe non avvenire correttamente.
Mi date una mano?
Grazie
Immagine:
784,24 KB
|
|
|
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 24 luglio 2018 : 20:38:09 Inserito il - 24 luglio 2018 : 20:38:09




|
La tua risposta mi sembra corretta (dici che le bande non si vedono benissimo, in realtà questo è un ottimo risultato reale, non quelli didattici)
Citazione:
Inoltre chiede il fenotipo nel caso in cui lo SNP cada in un introne.
Non "nel caso in cui lo SPN cada in un introne", ma lo SNP è su un sito accettore di splicing. Come è fatto e a cosa serve normalmente un sito accettore di splicing? Cosa accade se sostituisci la G con la A? Rispondi a queste domande e avrai risposto...
 |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
Fricerca
Nuovo Arrivato

3 Messaggi |
 Inserito il - 24 luglio 2018 : 21:25:53 Inserito il - 24 luglio 2018 : 21:25:53



|
Ciao domi84, innanzitutto grazie mille per la tua risposta :)
Io credo che se lo SNP cade in un introne, allora non sarà riconosciuto più come sito accettore di splicing.
Tuttavia i pazienti che hanno l'allele con lo SNP sono eterozigoti, quindi hanno un allele normale ed il loro fenotipo dovrebbe essere comunque wild-type.
Giusto? |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
|
|
Fricerca
Nuovo Arrivato

3 Messaggi |
 Inserito il - 25 luglio 2018 : 09:25:13 Inserito il - 25 luglio 2018 : 09:25:13



|
Grazie mille 
Buona giornata  |
 |
|
| |
Discussione |
|





