| Autore |
Discussione |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 23 novembre 2009 : 12:12:34 Inserito il - 23 novembre 2009 : 12:12:34




|
|
Salve!Avrei bisogno di un aiuto. Sapete mica dirmi per cortesia cosa si intende per primer intronspanning? Ringrazio in anticipo chiunque sappia dirmi qualcosa...buona giornata!
|
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 24 novembre 2009 : 16:41:40 Inserito il - 24 novembre 2009 : 16:41:40





|
Ciao,
a volte la risposta non arriva subito, ma bisogna attendere qualche giorno.
Però anche se non hai ottenuto una risposta per favore non reinserire la stessa discussione, anche se in una sezione diversa, perchè si genera solo confusione. Per questo motivo ho cancellato il tuo messaggio doppio.
Tornando alla tua domanda significa letteralmente "che si estende lungo l'introne" in pratica sono i primers disegnati a cavallo tra 2 esoni (la prima parte del primer alla fine di un esone e l'ultima parte all'inizio dell'esone successivo) in modo che si possono attaccare solo al cDNA e non al DNA genomico.
|
 |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 25 novembre 2009 : 14:22:17 Inserito il - 25 novembre 2009 : 14:22:17




|
| Grazie della risposta e mi scuso per aver inserito il messaggio anche nelle "tecniche", non voglio assolutamente creare della confusione. Grazie mille ancora e buona giornata! |
 |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 25 novembre 2009 : 14:39:00 Inserito il - 25 novembre 2009 : 14:39:00




|
| Vanno bene anche una coppia di primers in cui il forward è su un esone ed il reverse su un altro esone...?Grazie |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 25 novembre 2009 : 15:51:28 Inserito il - 25 novembre 2009 : 15:51:28





|
Vanno bene per cosa?
per funzionare in una PCR funzionano, ma non sono "intronspanning" (vista la definizione che ho detto) e possono amplificare anche DNA genomico che ha comunque gli esoni, l'unica differenza sarà la grandezza della banda: da cDNA avrai una banda di lunghezza inferiore (ci sono solo gli esoni compresi tra i primers) mentre per il gDNA avrai una banda più grande perchè sono compresi oltre agli esoni anche gli introni fra essi. |
 |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 25 novembre 2009 : 18:22:13 Inserito il - 25 novembre 2009 : 18:22:13




|
| Mi servono perchè devo estrarre l'RNA da cellule per poi caratterizzarle.Se nella PCR che faccio mi viene solo la banda della lunghezza dell'amplificato dovrebbero andare bene no?Il problema si porrebbe se mi uscisse anche una banda più alta, allora avrei contaminazione da DNA? Sto ragionando bene o no? Quando faccio l'estrazione uso la DNasi. Non sono ancora partita con il lavoro, ho iniziato però a vedere i primer che mi servono ma prima di ordinarli vorrei essere sicura...Grazie. |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 25 novembre 2009 : 19:08:28 Inserito il - 25 novembre 2009 : 19:08:28




|
Sì, andranno bene. Diciamo che se l'introne è piuttosto lungo non avrai probabilmente nemmeno amplificazione del genomico.
Tuttavia se vuoi andare sul sicuro al 100% (e se la sequenza te lo permette) fai almeno uno dei 2 primer a cavallo di due esoni. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 25 novembre 2009 : 22:52:36 Inserito il - 25 novembre 2009 : 22:52:36





|
| Beh se comunque fai il trattamento con DNasi non dovresti avere problemi, in ogni caso se fai una PCR qualitativa va benissimo, se invece vuoi fare una quantitativa sarebbe consigliabile che almeno uno dei primer sia a cavallo tra 2 esoni. Poi questo non vuol dire che tu non lo possa fare comunque. |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 26 novembre 2009 : 09:40:00 Inserito il - 26 novembre 2009 : 09:40:00




|
| Grazie. Scusate l'ignoranza....come faccio a costruire un primer intron spanning? Io i primer che ho scelto, gli ho scelti mediante il programma primer3. Sareste così gentili da spiegarmelo? Ora devo fare solo una qualitativa ma in futuro mi servirà anche scegliere dei primer per Real time. Grazie ancora. |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 26 novembre 2009 : 10:27:30 Inserito il - 26 novembre 2009 : 10:27:30





|
Primer 3 dovrebbe avere l'opzione, ma adesso non so dirti esattamente perchè in realtà non lo uso ma utilizzo altri programmi.
Comunque puoi utilizzare Primer BLAST (che tra l'altro per chi non lo sapesse utilizza Primer3) che oltre a disegnarti i primers ti fa anche il blast per testare la specificità.
È veramente facilissimo da utilizzare, comunque in "Exon/intron selection" trovi l'opzione "Primer must span an exon-exon junction".
In ogni caso anche se non metti questa quando vengono mostrati i risultati di dovrebbe dire quali primer sono all'interno di un esone e quali alla giunzione.
 |
 |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 26 novembre 2009 : 11:30:50 Inserito il - 26 novembre 2009 : 11:30:50




|
| Grazie, in giornata ci provo!Il BLAST comunque per i primer che avevo scelto lo avevo già fatto e mi diceva che i primer erano specifici per il mio gene. In ogni caso provo a cercare con questo programma i primer intron spanning perchè in questo modo sono più sicura!Ti faccio sapere... |
 |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 26 novembre 2009 : 16:20:59 Inserito il - 26 novembre 2009 : 16:20:59




|
Ciao! Avrei un pò di cose da chiederti...Sono andata su primerBLAST, ho inserito la sequenza presa da FASTA, ho messo l'opzione per i primer intron spanning e mi sono venute diverse paia di primer. Io ho scelto quella più corta, l'ultima della serie (203bp). Scegliere le ultime piuttosto che le prime è un problema? Le prime nominate sono le migliori?
Seconda domanda: ho verificato sia il forward che il reverse sulla sequenza da Ensemble, dove mi fa vedere sia gli introni che gli esoni e in effetti ho visto che entrambi sono a cavallo tra due esoni. Tra il forward ed il reverse c'è un introne lungo 1449bp. Se amplifico dna genomico dovrei accorgermene dalla presenza di una banda molto alta oltre alla mia di 203bp?
Terza domanda (scusa se stresso così tanto): il programma primerBLAST mi fa già il blast? Sono andata su Pubmed e su blast per verificare la specificità: ho trovato che la sequenza forward manca dell'ultima base nel gene che mi interessa (cioè sul cromosoma 12) mentre risulta completa nella sequenza di un altro gene sul cromosoma2. La sequenza del reverse non me la trova propio nel cromosoma 12 dove interessa a me, ma trova solo una parte della sequenza sul cromosoma 7.
Spero di essere stata chiara nell'esporre le mie domande. Grazie. |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 26 novembre 2009 : 18:13:08 Inserito il - 26 novembre 2009 : 18:13:08





|
mmm mi sa che non hai capito molto ce cos'è un primer intronspanning...
comunque vediamo se riesco a fare chiarezza.
Citazione:
Sono andata su primerBLAST, ho inserito la sequenza presa da FASTA, ho messo l'opzione per i primer intron spanning e mi sono venute diverse paia di primer. Io ho scelto quella più corta, l'ultima della serie (203bp). Scegliere le ultime piuttosto che le prime è un problema? Le prime nominate sono le migliori?
L'ordine non so bene con che criterio lo stabilisca, comunque dovrebbe essere lo stesso che hai in primer3 (essendo di fatto lo stesso programma), tutti i primers che ti dà vanno bene.
Se hai esigenze di avere l'amplificato di una certa grandezza puoi benissimo specificarlo in fase di ricerca.
Comunque poi poi analizzare anche i primers con altri programmi per vedere se fanno dimeri, strutture secondarie ecc...
Citazione:
Seconda domanda: ho verificato sia il forward che il reverse sulla sequenza da Ensemble, dove mi fa vedere sia gli introni che gli esoni e in effetti ho visto che entrambi sono a cavallo tra due esoni. Tra il forward ed il reverse c'è un introne lungo 1449bp. Se amplifico dna genomico dovrei accorgermene dalla presenza di una banda molto alta oltre alla mia di 203bp?
NO! Il DNA genomico non si amplifica! Che è appunto lo scopo di utilizzare primer a cavallo di giunzioni tra 2 esoni. Non può legarsi al DNA perchè sarebbe un pezzo di primer su un esone e un pezzo su quello successivo con l'introne in mezzo, quindi non si attacca!
Siccome la parte importante del primer per l'amplificazione è il 3', mentre al 5' volendo ci potresti inserire una coda che non si attacca al templato, ovviamente se del tuo primer di 20nt 1-2 si trovano sull'esone prima (5') e 18-19 sull'esone successivo (3') si può attaccare anche al DNA e amplificarlo.
Ma il programma evita di disegnarti primers del genere (e tra l'altro puoi anche scegliere il n. di basi minime che vuoi che siano su un esone o sull'altro).
Citazione:
Terza domanda (scusa se stresso così tanto): il programma primerBLAST mi fa già il blast?
certo dal nome: "primerBLAST" e lo scopo di utilizzarlo è quello, altrimenti sarebbe una copia di primer3.
Praticamente funziona così:
- usa primer3 per disegnare i primers
- fa il BLAST
cosa che prima andava fatta separatamente.
Citazione:
Sono andata su Pubmed e su blast per verificare la specificità: ho trovato che la sequenza forward manca dell'ultima base nel gene che mi interessa (cioè sul cromosoma 12) mentre risulta completa nella sequenza di un altro gene sul cromosoma2. La sequenza del reverse non me la trova propio nel cromosoma 12 dove interessa a me, ma trova solo una parte della sequenza sul cromosoma 7.
Beh è normale che non ti trovi le sequenze sui cromosomi che sono DNA, visto che è stata disegnata apposta per funzionare sull'RNA.
Poi se comunque hai la stessa sequenza in un altro cromosoma, ma solo di un primers ovviamente il prodotto aspecifico non si amplificherà.
Comunque il risultato di primer blast, preso da questo esempio che avevo fatto: http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=14920
è così:
Innanzitutto ti dovrebbe dire:
Specificity of primers Primer pairs are specific to input template as no other targets were found in selected database: NCBI Transcript Reference Sequences (Organism limited to Homo sapiens)
poi hai uno schemino di questo tipo:
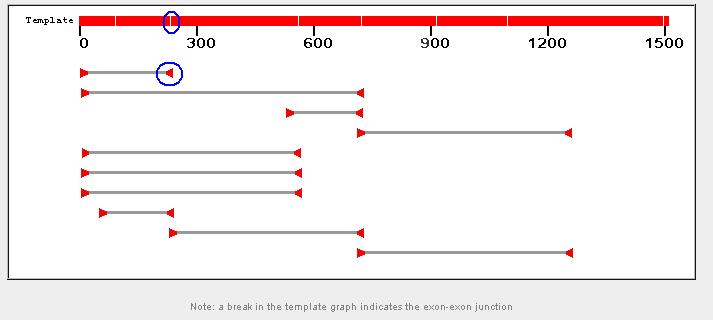
e come ti dice, le giunzioni tra gli esoni sono le righine bianche.
Ti ho segnato con un cerchietto blu il primer della prima copia che è a cavallo di una giunzione.
Il risultato di quella copia è questo:
Primer pair 1
Sequence (5'->3') Strand Length Start Stop Tm GC%
on template
Forward primer CAGCGGACAGCGCCAAGTGA Plus 20 7 26 59.98 65.00%
Reverse primer CCATGACGCCCTGGTGTCGG Minus 20 244 225 59.77 70.00%
Internal oligo Plus
Product length 238
Product Tm
Product Tm - min(OLIGO Tm)
Exon junction 234/235 (reverse primer) on template NM_001100.3
Total intron size
Products on intended target
>NM_001100.3 Homo sapiens actin, alpha 1, skeletal muscle (ACTA1), mRNA
product length = 238
Forward primer 1 CAGCGGACAGCGCCAAGTGA 20
Template 7 .................... 26
Reverse primer 1 CCATGACGCCCTGGTGTCGG 20
Template 244 .................... 225
in questo caso il reverse è a cavallo di una giunzione, se fai un blast su DNA non lo trovi. |
 |
|
|
lora
Nuovo Arrivato

52 Messaggi |
 Inserito il - 27 novembre 2009 : 10:26:04 Inserito il - 27 novembre 2009 : 10:26:04




|
| Grazie mille per avermi dato una risposta così dettagliata...finalmente mi sono chiarita le idee...sei stata davvero molto gentile, ora mi metterò a scegliere i primers in base a questi criteri!Buona giornata |
 |
|
|
margi08
Nuovo Arrivato

2 Messaggi |
 Inserito il - 07 febbraio 2017 : 16:10:30 Inserito il - 07 febbraio 2017 : 16:10:30



|
| ciao, io vorrei progettare dei primer che si attacchino alle porzioni introniche a destra e sinistra del mio esone. Non ho capito la tua affermazione: perchè si possono attaccare solo a cDNA e non a DNA genomico? |
 |
|
| |
Discussione |
|





