Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
ilchimicomat
Nuovo Arrivato

Prov.: Latina
Città: Aprilia

12 Messaggi |
 Inserito il - 27 febbraio 2009 : 10:27:43 Inserito il - 27 febbraio 2009 : 10:27:43





|
Salve ragazzi volevo chiedere se qualcuno di voi poteva aiutarmi a trovare la soluzione ad un quesito del mio prossimo esame di biologia molecolare sull'RNA.
La domanda centrale sulle tecniche è: come faccio ad individuare un interazione tra un miRNA e un gene?
Premetto che non ho intenzione di chiedere protocolli o altro, ma è sufficente esporre una logica di approccio, senza troppi tecnicismi. Io ho pensato alla seguente cosa:
- in vitro, prendo provetta con plasmide di espressione che porti la sequenza del gene che studio. Lo metto assieme all'apparato trascrizionale e ai fattori di capping, splicing e poliadenilazione. Ottengo così in provetta l'mRNA maturo del gene.
- prendo questo mRNA maturo e lo marco alle estremità, faccio la Southern e vedo quando pesa (in una provetta)
- in un'altra provetta inserisco i vari miRNA sintetizzati marcati anche loro, e come se fossero una sonda, questi si andranno ad ancorare alle loro sequenze complementari sul mRNA del gene. Faccio l'elettroforesi e avrò un ritardo nella corsa, seppur minimo (lo potrei amplificare?) Così capisco che il miRNA ha interagito con il mio gene.
Secondo voi questa sequenza di operazioni (molto fantasiose) potrebbe funzionare?
Se invece vorrei identificare la regione di mRNA precisa potrei aggiungere il complesso RISC+miRNA, digerire con RNAsi e poi fare la corsa per vedere l'impronta che resta? (tipo esperimento con micrococcale sui nucleosomi?)
Grazie per le eventuali risposte...
|
|
|
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 01 marzo 2009 : 14:25:26 Inserito il - 01 marzo 2009 : 14:25:26





|
Secondo me sei andato a impelagarti in giri di operazioni
laboriosissime e a tratti impossibili da mettere in pratica
quando invece la soluzione è dannatamente più semplice.
Citazione:
- in vitro, prendo provetta con plasmide di espressione che porti la sequenza del gene che studio. Lo metto assieme all'apparato trascrizionale e ai fattori di capping, splicing e poliadenilazione. Ottengo così in provetta l'mRNA maturo del gene.
Ammesso che sia possibile una roba così complicata
(francamente non credo, almeno per quanto riguarda lo
splicing in vitro) non faresti prima a fare una semplice
trascrizione in vitro con i metodi tradizionali?
Ossia: cDNA (nessun problema di introni), costrutto
con promotore T7 (nessun problema di cofattori trascrizionali)
Non avresti il cap e la poly-A,ok, ma tanto in vitro che te ne fai?
Citazione:
prendo questo mRNA maturo e lo marco alle estremità, faccio la Southern e vedo quando pesa (in una provetta)
Prova a dire una roba del genere all'esame di biologia
molecolare e una botola si aprirà inesorabilmente sotto di te.
Nozione base: il southern si fa con il DNA, il nothern con l'RNA
Citazione:
in un'altra provetta inserisco i vari miRNA sintetizzati marcati anche loro, e come se fossero una sonda, questi si andranno ad ancorare alle loro sequenze complementari sul mRNA del gene. Faccio l'elettroforesi e avrò un ritardo nella corsa, seppur minimo (lo potrei amplificare?) Così capisco che il miRNA ha interagito con il mio gene.
Un EMSA con l'RNA non so se si possa fare, non credo (ma non lo
escludo) perchè secondo me sarebbe difficilissimo impedire la
degradazione dell'RNA, salvo trattare con formamide o quant'altro,
ma così il tuo base-pairing ne risente.
Ma soprattutto dovresti domandarti: che informazioni
"biologiche" ti può dare un esperimento così??
Farlo in vivo ti richiederebbe meno tempo e risultati più informativi!
Semplicemente:
Overesprimi il tuo miRNA putativo in cellule transfettate
con il tuo gene e ne controlli l'espressione (a livello proteico,
ovviamente, visto che i miRNA di solito non degradano l'mRNA).
Citazione:
Se invece vorrei (!!!!!!!VOLESSI!!!!!!!) identificare la regione di mRNA precisa potrei aggiungere il complesso RISC+miRNA, digerire con RNAsi e poi fare la corsa per vedere l'impronta che resta? (tipo esperimento con micrococcale sui nucleosomi?)
Anche qui: il fingerprint è un tecnica che si usa per il DNA,
non so se si possa usare anche per l'RNA, senza contare che
in vitro non avrai mai TUTTI i fattori del complesso che si
assembla in vivo (chissà quanti sono in realtà) e quindi avrai
informazioni parziali, incomplete e comunque poco affidabili
quando invece potresti ottenerla molto, molto, più facilmente.
Che bisogno c'è di fare tutto questo?
Pensaci un secondo :
quando un miRNA si appaia su un mRNA,
in QUALI REGIONI si appaia, e PERCHE' ??
Se hai studiato dovresti saperlo, non ti resta che fare 2+2
|
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
ilchimicomat
Nuovo Arrivato

Prov.: Latina
Città: Aprilia

12 Messaggi |
 Inserito il - 02 marzo 2009 : 12:00:32 Inserito il - 02 marzo 2009 : 12:00:32





|
Allora innanzitutto grazie della risposta che è utilissima e mi permette di capire moltissime cose. Premesso che sono un novizio delle biotecnologie, infatti sto solo al secondo anno e questo semestre abbiamo incominciato molecolare, ti dò alcune mie osservazioni in merito alle tue risposte:
a- alcuni miRNA sono prodotti da seq. introniche ---> escludo a priori cDNA
b- il promotore virale forte è scontato nel plasmide di espressione
c- southern/northen è un errore inqualificabile ahimè, mi confondo sempre
d- il tuo "overesprimi e contolli a livello proteico" intende il fatto che utilizzando questo approccio genetico posso vedere gli effetti che ottengo? Ma ciò non è molto pratico se il gene con cui interagisce il mio miRNA non è ancora noto, o dà una risposta molto evidente, no?
e- se in vivo faccio l'ultimo punto (quello della sequenza precisa) il complesso RISC lo ho tutto e sapendo che i miRNA legano le 3'-UTR degli mRNA che ci faccio? IL 2+2 non sono riuscito a completarlo...aiutino?
P.S. Come vedi le mie conoscenze non sono approfondite proprio dal fatto che sono un novizio e che cmq non è un argomento affrontato in maniera completa ed esauriente dal prof...Grazie cmq sei utilissimo  |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 02 marzo 2009 : 13:15:05 Inserito il - 02 marzo 2009 : 13:15:05




|
Faccio una piccola precisazione: fare l'esperimento in cellule è sempre considerato in vitro.
In vivo vuol dire farlo in un animale. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 02 marzo 2009 : 16:00:16 Inserito il - 02 marzo 2009 : 16:00:16





|
Citazione:
Messaggio inserito da ilchimicomat
d- il tuo "overesprimi e contolli a livello proteico" intende il fatto che utilizzando questo approccio genetico posso vedere gli effetti che ottengo? Ma ciò non è molto pratico se il gene con cui interagisce il mio miRNA non è ancora noto, o dà una risposta molto evidente, no?
do pienamente ragione a dioniso
l'attività dei miRNA è a livello post-trascrizionale,inibiscono i fenomeni di traduzione del messaggero,per cui basta che fai esprimere il tuo gene di interesse in assenza dei miRNA e poi rifai la stessa cosa ma aggiungi miRNA nella trua provetta e vedi se c è ancora espressione.
da quello che ho letto mi pare di capire che te sei preoccupato dal fatto che nn conosci l'attività del gene targhet;ma non ti deve fregare della tua funzione,per verificare l'attività di un miRNA ti basta sapere se in sua presenza c è espressione del gene o non c è.
PER INDIVIDUARE LA REGIONE VOLEVO ChIEDERE SE INVECE si poteva sfruttare la tecnica dell' RNA-protection. |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 02 marzo 2009 : 20:03:05 Inserito il - 02 marzo 2009 : 20:03:05





|
Il messaggio di fondo è semplicemente questo: la dimostrazione "in provetta" di un appaiamento
tra un trascritto e un miRNA non vale assolutamente niente da un punto di vista biologico.
Se tu vuoi dimostrare che un "gene è BERSAGLIO di un miRNA" (perchè un gene non interagisce
direttamente con un microRNA, è semmai il suo mRNA che interagisce con un microRNA,
giusto?) non basta dire che i due si appaiano, perchè questa è una cosa che puoi verificare
con un certo grado di certezza già solo conoscendo le sequenze. Siccome sai che i miRNA
animali si appaiano nel 3'UTR mediante al loro sequenza "seed" - aiutandosi eventualmente
con altre basi all'estremità opposta- se conosci la sequenza del tuo trascritto - comprese UTR -
e vuoi provare a vedere se un certo miRNA ci si appaia, basta fare un'analisi bioinformatica.
Già quello ti dice che POTREBBERO appaiarsi, ma non significa che uno sia il target dell'altro!
L'unico modo per avere questa informazione è vedere se in un sistema "vivente" (una
cellula in coltura) la presenza contemporanea e del trascritto del tuo gene e del miRNA che
tu ipotizzi possa regolarlo, porti ad un effettivo abbassamento della TRADUZIONE. Questo
significherebbe che il trascritto è bersaglio del miRNA. Non t'importa nulla della funzione
del tuo gene, questo esperimento lo puoi fare lo stesso né più e né meno. Basta essere in
grado di esprimere il gene in modo ricombinante e avere una metodica per dosarne il prodotto
proteico (devi avere un Ab per farci western blot, quello sì).
Certo, dopo che hai avuto i tuoi risultati biologici, se vedi che effettivamente il miRNA
riesce a downregolare il trascritto, volendo puoi anche fare un RNA protection assay per
vedere se si forma un double strand. GFPina - che la sa lunga ( )- mi ha pure fatto )- mi ha pure fatto
vedere che esistono degli EMSA per validare l'appaiamento , anche se non ho capito
bene come possa un miRNA di 22 nt ritardare la corsa di un trascritto di diverse kb
(magari pure in presenza di agenti denaturanti come la formamide/formaldeide che
ne inficiano il già precario base-pairing) Immagino ci sarà bisogno di fare qualche
aggiustamento al protocollo, tipo crosslinkare miRNA e mRNA e magari coniugare
il miRNA con un'ancora (una bead, forse) però non so, non ho mai visto
fare niente del genere e io non lo farei, anche se pare che qualcuno lo faccia.
Così facendo puoi confermare che effettivamente si forma l'appaiamento che puoi immaginare
guardando le sequenze al computer, e potrebbe essere un'info importante perchè l'RNA è
una molecola dinamica e gli appaiamenti/disappaiamenti possono essere traballanti e
non sempre ugualmente possibili (non è come col DNA). Però io per validare i risultati
non credo farei nulla del genere, ma piuttosto proverei a mutagenizzare la 3'UTR per
dimostrare che quando cambiano precise basi cambi anche la possibilità da parte del
miRNA di regolare l'espressione del target. Questo sarebbe una prova schiacciante.
|
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 02 marzo 2009 : 20:58:32 Inserito il - 02 marzo 2009 : 20:58:32





|
Piccola precisazione necessaria: quando dicevo "in vivo" intendevo in una
cellula vivente, per distinguere dalla provetta, che è "in vitro" in senso
stretto. Non intendevo in un animale, per cui ho usato un termine
un po' equivoco. Sorry. (thanx Nico :) ) |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
ilchimicomat
Nuovo Arrivato

Prov.: Latina
Città: Aprilia

12 Messaggi |
 Inserito il - 02 marzo 2009 : 21:52:51 Inserito il - 02 marzo 2009 : 21:52:51





|
Grazie Dyonisos, mi hai aperto la mente, anche se dovendo essere onesto molti termini da voi usati per me sono molto ma molto IGNOTI (tipo Ab, bead, crosslinkare ecc...) Vabbe li apprenderò con l'esperienza.
Oggi pomeriggio ho riflettuto sul protocollo che mi avevi aiutato a scrivere correggendo le mie prime opinioni e anche con le ultime aggiunte che mi hai fornito nell'ultimo intervento sono arrivato alla conclusione che:
- prendo una coltura di lieviti mutanti per il gene codificante il miRNA che voglio studiare, osservando così il fenotipo (se ne ottengo uno) che ho in piastra del gene sovraespresso
- prendo un'altra coltura dove inserisco tramite plasmide di espressione (promotore virale + seq del miRNA) la sequenza del miRNA che intendo studiare. Ottengo così una attenuazione della traduzione e correlo l'effetto con quello osservato precedentemente così ricavo che il miRNA regola quel gene in particolare
- infine mutagenizzo la sequenza 3'-UTR del gene in questione e vedo se il miRNA ci si lega lo stesso e se si che effetto ha.
Ho riscritto i passi correttamente? Spero la logica questa volta sia corretta e "realizzabile" anche se purtroppo non avendo delle forti basi molecolari e sperimentali alle spalle non posso abbinare le tecniche corrette. Spero che ora vada bene. Se hai altri suggerimenti postali pure. Grazie mille
P.S. Mi ero dimenticato di una cosa. Ma se il mio gene con il miRNA non mi produce nessun fenotipo visibile, come faccio a vedere l'espressione proteica? Non penso che si faccia un Western di tutta la proteomica di quella cellula no? |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 02 marzo 2009 : 22:36:02 Inserito il - 02 marzo 2009 : 22:36:02




|
Citazione:
Grazie Dyonisos, mi hai aperto la mente, anche se dovendo essere onesto molti termini da voi usati per me sono molto ma molto IGNOTI (tipo Ab, bead, crosslinkare ecc...) Vabbe li apprenderò con l'esperienza.
Ab = anticorpo, dall'inglese antibody
bead = biglia, sfera (in questo contesto di dimensioni microscopiche ovviamente)
crosslinkare = italianizzazione dell'inglese "to cross-link", cioè "fare dei legami crociati".
Citazione:
P.S. Mi ero dimenticato di una cosa. Ma se il mio gene con il miRNA non mi produce nessun fenotipo visibile, come faccio a vedere l'espressione proteica? Non penso che si faccia un Western di tutta la proteomica di quella cellula no?
Non ho letto con attenzione quanto scritto sopra, ma la tua domanda è: il miRNA interagisce con il gene X?
Benissimo, il gene X produrrà una proteina, ed è su quella che vai a fare il Western blot.
Se non hai un anticorpo per la proteina codificata da X ti dovrai arrangiare in altri modi (suppongo che nel momento in cui fai un lavoro del genere tu abbia abbastanza informazioni su questa proteina ed abbia anche un qualsiasi metodo per determinarne la presenza) |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 03 marzo 2009 : 00:31:30 Inserito il - 03 marzo 2009 : 00:31:30





|
Citazione:
Messaggio inserito da Dionysos
... GFPina - che la sa lunga ( )- mi ha pure fatto vedere che esistono degli EMSA per validare l'appaiamento , anche se non ho capito bene come possa un miRNA di 22 nt ritardare la corsa di un trascritto di diverse kb (magari pure in presenza di agenti denaturanti come la formamide/formaldeide che ne inficiano il già precario base-pairing) Immagino ci sarà bisogno di fare qualche aggiustamento al protocollo, tipo crosslinkare miRNA e mRNA e magari coniugare il miRNA con un'ancora (una bead, forse) però non so, non ho mai visto fare niente del genere e io non lo farei, anche se pare che qualcuno lo faccia. )- mi ha pure fatto vedere che esistono degli EMSA per validare l'appaiamento , anche se non ho capito bene come possa un miRNA di 22 nt ritardare la corsa di un trascritto di diverse kb (magari pure in presenza di agenti denaturanti come la formamide/formaldeide che ne inficiano il già precario base-pairing) Immagino ci sarà bisogno di fare qualche aggiustamento al protocollo, tipo crosslinkare miRNA e mRNA e magari coniugare il miRNA con un'ancora (una bead, forse) però non so, non ho mai visto fare niente del genere e io non lo farei, anche se pare che qualcuno lo faccia.
Innanzitutto visto che sono stata tirata in ballo  , confermo che l'EMSA si può fare, non posso inserire immagini visto che tutti gli articoli che ho trovato al riguardo non sono free. Però ho fatto uno schemino: , confermo che l'EMSA si può fare, non posso inserire immagini visto che tutti gli articoli che ho trovato al riguardo non sono free. Però ho fatto uno schemino:
Si effettua una trascrizione in vitro dell'RNA X che si vuole analizzare, o solo del suo 3'UTR. Si marca il miRNA Y che si pensa interagisca con l'RNA X. Si mettono assieme nella stessa provetta e se interagiscono si legheranno. Se vengano crosslinkati non lo so (dovrei cercare un protocollo dettagliato). Comunque poi si fanno migrare su un gel di poliaclimade nativo. Se c'è interazione si osserverà una banda a PM maggiore, data dal miRNA + (3'UTR)-RNA X.
In basso si vedrà il miRNA non legato, che migra al suo PM, circa 22bp.
Come ulteriore prova si mutagenizza il 3'UTR nella sequenza che si pensa legare il mi-RNA e si procede allo stesso modo. Si osserverà solo la banda del miRNA a 22bp e non la banda "ritardata" perchè non c'è più interazione.
Penso sia chiaro a questo punto Dionysos, che non è il miRNA che ritarda la corsa del trascritto, ma vice versa  , per questo lo vedi su gel. , per questo lo vedi su gel.
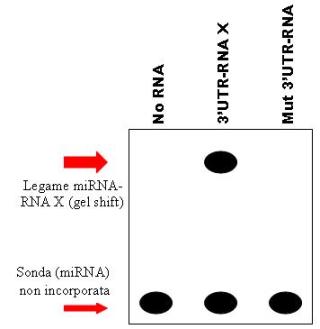
Comunque ilchimicomat sinceramente non ho capito da dove vuoi partire e cosa vuoi fare.
Se dici "come faccio ad individuare un interazione tra un miRNA e un gene?"
(a parte la puntualizzazione che è stata fatta che il miRNA interagisce con l'RNA e non con il DNA)
io presuppongo che tu intenda che hai un certo miRNA y e vuoi vedere se interagisce con l'RNA X.
Se invece non hai ancora individuato l'RNA X con cui il miRNA Y potrebbe interagire il discorso è completamente diverso. Dovresti almeno chiarire questo punto prima di passare ai possibili esperimenti!
I lieviti poi... li utilizzi se stai studiando un RNA/proteina di lievito. Ma del "fenotipo" non ti interessa molto, devi solo dimostrare che miRNA e RNA interagiscono.
Il meccanismo è semplice:
miRNA Y + RNA X --> blocco della traduzione di X --> non hai proteina X
quindi tu vai a vedere se hai proteina X (con Western Blot come ti è stato detto prima), ma la funzione di X non ti interessa in questo caso.
Questa è la base, poi ti puoi inventare i vari esperimenti: Mutagenizzare il 3'UTR dell'RNA X, overesprimere il miRNA Y, silenziare l'RNA X e vedere se hai gli stessi effetti che hai con l'overespressione del miRNA Y, inibire il miRNA Y endogeno... ecc |
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 03 marzo 2009 : 00:41:40 Inserito il - 03 marzo 2009 : 00:41:40





|
Citazione:
Messaggio inserito da chick80
Non ho letto con attenzione quanto scritto sopra, ma la tua domanda è: il miRNA interagisce con il gene X?
Benissimo, il gene X produrrà una proteina, ed è su quella che vai a fare il Western blot.
Se non hai un anticorpo per la proteina codificata da X ti dovrai arrangiare in altri modi (suppongo che nel momento in cui fai un lavoro del genere tu abbia abbastanza informazioni su questa proteina ed abbia anche un qualsiasi metodo per determinarne la presenza)
mmm,interessante,e se effettivamente non ho questo benedetto anticorpo perchè non conosco la proteina targhet (vattela a pesca il il perchè)potrei introdurre nel cell-free un eccesso di aa marcati radioattivamente,per rilevare la proteina sul gel??oppure questi aa mi vengono incorporati anche altrove,cioè nei sistemi cell-free avviene solo la traduzione del nostro mRNA di interesse,o anche di altri mRNA di cui nn ci frega nulla? |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 03 marzo 2009 : 00:45:06 Inserito il - 03 marzo 2009 : 00:45:06





|
C'è un piccolo problemino :D... i lieviti non hanno la pathway dell'RNA interference!!!!
(hai scelto forse l'unico organismo eucariotico in cui non puoi fare l'esperimento :P )
Tutto quello che hai scritto va bene, solo devi farlo in cellule eucariotiche
che esprimano tutti quei fattori necessari alla pathway dei microRNA (sono sicuro
che me li saprai elencare tutti!! :D).
Una cosa molto importante che hai scritto è il problema del fenotipo: non è un
problemino da poco, anzi è il motivo per cui i miRNA non sono stati "scoperti"
prima (diciamo che non è stato scoperto il loro ruolo di regolatori dell'espress. genica).
Infatti la regolazione imposta dai miRNA è molto sottile e promiscua: possono regolare
diversi geni contemporaneamente, senza reprimerli pesantemente e comunque in modo diverso
a seconda dell'affinità di appaiamento. Il risultato è che un fenotipo miRNA knockout spesso
non ha nessun chiaro carattere "osservabile" e non è facile analizzarlo a livello molecolare
con le metodiche tradizionali (ci si è riusciti solo per let-7 di C. elegans e pochi altri)
ecco eprchè prima dell'avvento delle tecnologie post-genomiche non è stato possibile individuare
questi attori molecolari. Un fenotipo miRNA può essere descritto accuratamente ed in
modo completo solamente attraverso un profiling del cosiddetto "protein output" (un'analisi
proteomica , sostanzialmente) altrimenti si osservano solo CERTI ASPETTI del fenotipo.
Quindi tornando coi piedi per terra: se tu vuoi controllare se un miRNA influenza
(tra i tanti) anche l'espressione del TUO gene, devi avere la possibilità di esprimere
il TUO gene in modo ricombinante e misurare la quantità di proteina espressa in qualche modo.
|
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 03 marzo 2009 : 00:53:50 Inserito il - 03 marzo 2009 : 00:53:50





|
Uh ma intanto mi ha risposto GFPina!!
non pensavo si potesse fare una sonda per un miRNA :/
però in effetti ha senso 
Mi è venuta in mente una cosa: si è parlato fare KO per i miRNA.
Il problema è che fare KO per i mIRNA non è uno scherzo, spesso i geni sono disposti in
clusters oppure non sono stati neppure mappati. Esistono metodi complessi per fare knock
down post-trascrizionale del miRNA (si usano vettori lentivirali "miRNA sponges", come
delle spugne che sequestrano uno specifico miRNA) che però funzionano abbastanza bene
ma per l'esperimento di cui parlavamo, ribadisco e arciribadisco, la cosa migliore
da fare è OVERESPRIMERE lo specifico miRNA in presenza del trascritto target putativo. |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 03 marzo 2009 : 00:56:31 Inserito il - 03 marzo 2009 : 00:56:31





|
Citazione:
Messaggio inserito da Dionysos
C'è un piccolo problemino :D... i lieviti non hanno la pathway dell'RNA interference!!!!
(hai scelto forse l'unico organismo eucariotico in cui non puoi fare l'esperimento :P
ehm..... Dionyso sbarelli??
i lieviti ce l hanno il pathway,tantè chè nel S.Pombe SI è VISTO PROPRIO che i miRNA comportano una modificazione epigenetica.Se non vado errato i mutanti dicer e argonauta di S.pombe mancano di una metilazione su una lisina dell'istone H3 che è un segnale coinvolto per l'interazione del centromero con la coesina ecc.
com è sta storia che nn hanno patwhay,magari mi confondo io? |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 03 marzo 2009 : 01:04:37 Inserito il - 03 marzo 2009 : 01:04:37





|
Citazione:
Messaggio inserito da zerhos
mmm,interessante,e se effettivamente non ho questo benedetto anticorpo perchè non conosco la proteina targhet (vattela a pesca il il perchè)potrei introdurre nel cell-free un eccesso di aa marcati radioattivamente,per rilevare la proteina sul gel??oppure questi aa mi vengono incorporati anche altrove,cioè nei sistemi cell-free avviene solo la traduzione del nostro mRNA di interesse,o anche di altri mRNA di cui nn ci frega nulla?
Non ho capito assolutamente il punto di quello che hai scritto.
Comunque in sistemi cell free se metti il vettore con il tuo mRNA X hai la traduzione solo di quello, non di altri mRNA (da dove li prenderebbero se non glieli metti tu?)
Poi una volta che hai la tua proteina marcata, cosa te ne fai?
Qua si stava parlando di interazione tra miRNA e RNA, non dell'identificazione di una proteina conoscendo l'RNA. |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 03 marzo 2009 : 01:08:51 Inserito il - 03 marzo 2009 : 01:08:51





|
Se sbarello o no te lo saprò dire dopo che mi avrai
indicato un articolo in cui si parla di RNA interference
(che è un fenomeno post-trascrizionale) nei lieviti.
Io, decisamente, devo essermelo perso : ) |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
|
|
zerhos
Utente Junior


Prov.: Pisa
Città: Pisa

421 Messaggi |
 Inserito il - 03 marzo 2009 : 01:28:46 Inserito il - 03 marzo 2009 : 01:28:46





|
Citazione:
Messaggio inserito da GFPina
Citazione:
Messaggio inserito da zerhos
mmm,interessante,e se effettivamente non ho questo benedetto anticorpo perchè non conosco la proteina targhet (vattela a pesca il il perchè)potrei introdurre nel cell-free un eccesso di aa marcati radioattivamente,per rilevare la proteina sul gel??oppure questi aa mi vengono incorporati anche altrove,cioè nei sistemi cell-free avviene solo la traduzione del nostro mRNA di interesse,o anche di altri mRNA di cui nn ci frega nulla?
Non ho capito assolutamente il punto di quello che hai scritto.
Comunque in sistemi cell free se metti il vettore con il tuo mRNA X hai la traduzione solo di quello, non di altri mRNA (da dove li prenderebbero se non glieli metti tu?)
Poi una volta che hai la tua proteina marcata, cosa te ne fai?
Qua si stava parlando di interazione tra miRNA e RNA, non dell'identificazione di una proteina conoscendo l'RNA.
sisi GFPina hai perfettamente ragione,la mia domanda era solo fine a se stessa,ma nn l ho puntualizzato,volevo solo sapere se nn si aveva un anticorpo per individuarla sulla corsa come si poteva procedere,esulando dal discorso dell'miRNA. |
"l'unica differenza tra me e un pazzo è che io non sono pazzo!"
Salvador.Dalì
|
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 03 marzo 2009 : 01:35:15 Inserito il - 03 marzo 2009 : 01:35:15





|
E' molto interessante, non conoscevo questi lavori, ma devo dire
ma non c'entrano nulla con i meccanismi di RNAi che conosciamo.
Qui non si parla di regolazione dell'espressione di geni mediante
RNA intereference: qui si parla di "heterocromatin silencing" che
è un fenomeno nucleare di silenziamento epigenetico, facente parte
del più vasto gruppo dei fenomeni di RNA silencing.
L'RNAi è una pathway nucleare e citoplasmatica conservata a partire
dagli Invertebrati (C. elegans e D. melanogaster), infatti pare che
nei lieviti - anche solo leggendo l'abstract che hai indicato -
non ci siano tutte le componenti necessarie per metterlo in atto,
ma solo dei geni omologhi ancestrali con ruoli nell'epigenetica.
Poi si possono fare un mare di chiacchiere:
innanzitutto se non c'è l'azione di RISC come si fa a parlare di RNA interference?
Gli autori dell'articolo propongono una speculazione del termine, ma non significa
che la cosa sia ancora scientificamente accettata. Potrebbe diventarlo. Chi lo sa.
Al di là delle speculazione terminologiche: nei lieviti non ho ancora visto
fare un esperimento di RNA interference per l'espressione genica! |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 03 marzo 2009 : 08:17:32 Inserito il - 03 marzo 2009 : 08:17:32




|
Citazione:
mmm,interessante,e se effettivamente non ho questo benedetto anticorpo perchè non conosco la proteina targhet (vattela a pesca il il perchè)potrei introdurre nel cell-free un eccesso di aa marcati radioattivamente,per rilevare la proteina sul gel??oppure questi aa mi vengono incorporati anche altrove,cioè nei sistemi cell-free avviene solo la traduzione del nostro mRNA di interesse,o anche di altri mRNA di cui nn ci frega nulla?
No, no, niente radioattivo... anche perchè verrebbe appunto incorporato in tutti gli mRNA.
Se la proteina ha attività enzimatica potresti fare un saggio sulla quantità di substrato consumato o prodotto generato.
Oppure si potrebbe mettere un tag alla proteina e rilevare quel tag.
Se la proteina è secreta si può mettere un tag fluorescente e valutare la fluorescenza del perfusato.
O magari hai un Ab ma non funziona in Western, però funziona in un qualche altro saggio (es. IIC, ELISA).
Insomma, poi dipende un po' dalla situazione.
Ribadisco, non credo uno si vada a impelagare in uno studio del genere se non sa nulla del gene che sta studiando... |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
ilchimicomat
Nuovo Arrivato

Prov.: Latina
Città: Aprilia

12 Messaggi |
 Inserito il - 03 marzo 2009 : 11:30:31 Inserito il - 03 marzo 2009 : 11:30:31





|
Wow ho visto che la discussione che ho iniziato si è notevolemente espansa. Innanzitutto grazie a tutti per l'interessamento e per le risposte che mi state continuamente dando.
Preciso che purtroppo la mia domanda iniziale è così come era nel compito di molecolare dell'appello precedente, ne più ne meno. Il professore infatti con la sua scarnità di informazioni voleva esclusivamente che noi sviluppassimo un po di "fantasia" nell'approcciare un problema reale. Quindi non posso fornirvi più informazioni di queste.
Detto ciò quoto alla grande GFPina (grazie!!!) che ha praticamente dato supporto tecnico alle mie primarie illazioni, dette senza molto fondamento nel primo post. Ora capisco che in fondo un pochino di verità ero riuscito a tirarla fuori dal mio cervello. Di fatto dunque dopo il disegno illustrativo postatomi in precedenza, ho capito e la dò come una risposta definitiva (almeno credo?!?!) quello che intendo fare:
- trascrizione in vitro del gene X da cui ricavo l'mRNA X
- sintesi del microRNA Y e marcatura terminale
- in vitro --> EMSA (il gel di cui mi parlavi come si chiama? nn è poliacrilammide?)
- risultati convalidati attraverso confronto con mutagenti delle 3'-UTR del gene (no legame, no ritardo)
Se poi devo attestare risultati biologici (boh?!? dipende da come gira al prof) metto in pratica il protocollo di Dyonisos:
- overespressione del gene X --> western blotting e controllo proteico
- introduzione del miRNA marcato nel lievito ---> western e controllo proteico (poca traduzione)
- conclusioni convalidate con mutanti 3'-UTR (per verificare l'effettivo legame all'mRNA X)
Spero che questi due protocolli possano andare bene. Se avete altre interessanti novità a questo punto sono affascinato nell'ascoltarvi  ... ... |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 03 marzo 2009 : 14:21:06 Inserito il - 03 marzo 2009 : 14:21:06





|
Citazione:
Messaggio inserito da ilchimicomat
Preciso che purtroppo la mia domanda iniziale è così come era nel compito di molecolare dell'appello precedente, ne più ne meno. Il professore infatti con la sua scarnità di informazioni voleva esclusivamente che noi sviluppassimo un po di "fantasia" nell'approcciare un problema reale. Quindi non posso fornirvi più informazioni di queste.
Beh allora penso che intendesse proprio come dimostri che c'è interazione tra miRNA e RNA.
Citazione:
Messaggio inserito da ilchimicomat
(il gel di cui mi parlavi come si chiama? nn è poliacrilammide?)
Si è poliacrilammde, ho sbagliato a scrivere! (errore di stompa  ) )
Citazione:
Messaggio inserito da ilchimicomat
Se poi devo attestare risultati biologici (boh?!? dipende da come gira al prof) metto in pratica il protocollo di Dyonisos:
- overespressione del gene X --> western blotting e controllo proteico
- introduzione del miRNA marcato nel lievito ---> western e controllo proteico (poca traduzione)
- conclusioni convalidate con mutanti 3'-UTR (per verificare l'effettivo legame all'mRNA X)
Non mi sembrava fosse così quello che ha detto Dionysos!
Se fai overesprimere il gene X vedi che aumenta la proteina X... ma questo è ovvio, non ha senso come esperimento!
In Lievito si stava discutendo che non ci sono miRNA quindi fai tutto in cellule eucariotiche.
Comunque a cosa ti servirebbe mettere un miRNA marcato? a cosa ti serve la marcatura?
Tu nella tua cellula hai già l'RNA X e il miRNA che si lega.
Quello che puoi fare è overesprimere il miRNA, in questo modo il miRNA si legherà all'RNA X inibendone la traduzione, quindi fai un western blot e vedi che c'è meno proteina rispetto al controllo (cellule non transfettate).
Poi come ti dicevo puoi fare l'opposto: inibendo il miRNA (trasfetti con un vettore miRNA antisenso) e dovresti vedere un aumento della proteina X.
La mutagenesi del sito 3'UTR si può fare, ma tieni conto che diventa più difficile perché nelle tue cellule avresti comunque l'RNA X con il 3'UTR wt. |
 |
|
|
ilchimicomat
Nuovo Arrivato

Prov.: Latina
Città: Aprilia

12 Messaggi |
 Inserito il - 03 marzo 2009 : 18:08:44 Inserito il - 03 marzo 2009 : 18:08:44





|
Nell'approccio biologico intendevo selezionare mutanti eucariotici che non abbiano la sequenza codificante per il miRNA nel loro genoma. Quindi facendo il western blotting dimostrerebbero un'ingente quantità di prodotto proteico in confronto a cellule eucariotiche che invece hanno il miRNA iniettato dentro. Cos' correlerei la presenza del micro che ci si lega e che regola la traduzione.
Poi ovviamente come fatto sopra, vado ad overesprimere il miRNA oppure mutagenizzo le 3'-UTR dell'mRNA X andando a ricombinare il gene X da cui deriva, e vedo che accade sia a livello proteico sia nella cellula in generale |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 04 marzo 2009 : 02:38:49 Inserito il - 04 marzo 2009 : 02:38:49





|
Citazione:
Messaggio inserito da ilchimicomat
Nell'approccio biologico intendevo selezionare mutanti eucariotici che non abbiano la sequenza codificante per il miRNA nel loro genoma.
Beh ecco è questo il punto! Non è come nei batteri o nel lievito che per caso nel terreno di coltura trovi dei mutanti!
Senza contare che, "come faresti ad isolarli"???
Un conto è isolare ad es un batterio resistente ad un certo antibiotico, metti l'antibiotico e quel ceppo crescerà, oppure un mutante auxotrofo...
Ma per isolare un mutante a cui manca quel miRNA, sempre ammesso che esista, come faresti?
Utilizzi delle cellule (di Eucarioti superiori) che in genere sono linee cellulari (o più difficilmente colture primarie), il "mutante" lo devi creare tu facendo un knock-out, e la cosa diventa più complessa.
Citazione:
Messaggio inserito da ilchimicomat
Quindi facendo il western blotting dimostrerebbero un'ingente quantità di prodotto proteico in confronto a cellule eucariotiche che invece hanno il miRNA iniettato dentro.
Mi sembra ci sia ancora confusione su questo punto, non è che le cellule eucariotiche hanno "il miRNA iniettato dentro", ce l'hanno dentro perché è endogeno! Esiste il gene che codifica per quel miRNA e il miRNA viene trascritto!
Quello che puoi fare come ti ho già detto è overesprimere il miRNA trasfettando (non "iniettando dentro" comunque) con un plasmide per l'espressione di quel miRNA.
Citazione:
Messaggio inserito da ilchimicomat
...oppure mutagenizzo le 3'-UTR dell'mRNA X andando a ricombinare il gene X da cui deriva
  
Questa non l'ho capita molto!
Io partirei da linee cellulari ko per quel determinato gene X (che se vuoi fare gli esperimenti di prima ti devi fare) e in queste transfetterei gli mRNA con le 3'-UTR mutagenizzate. |
 |
|
|
ilchimicomat
Nuovo Arrivato

Prov.: Latina
Città: Aprilia

12 Messaggi |
 Inserito il - 04 marzo 2009 : 10:08:47 Inserito il - 04 marzo 2009 : 10:08:47





|
L'isolamento, non avendo la vostra esperienza ovviamente è una questione solo di logica, non so se realmente si può fare.
Ho usato il termine "iniettato dentro" per differenziare le cellule mutanti da quelle normali (è ovvio che hanno il miRNA nel loro genoma, almeno quello)
Per mutagenizzare le 3'-UTR non posso operare direttamente sulle basi nucleotidiche del genoma e quindi ho pensato ci faccio inserire tramite trasposizione omologa un breve tratto di sequenza estranea che mi rompe la cornice di lettura corretta relativa alla 3', in modo tale che così viene alterata... |
 |
|
|
Garzgul
Utente Junior

Prov.: Roma
Città: Roma

183 Messaggi |
 Inserito il - 04 marzo 2009 : 19:39:07 Inserito il - 04 marzo 2009 : 19:39:07





|
Allora premetto che è il primo post che metto e anche io domani ho l'esame di Biologia Molecolare, dato che anche io sono al II anno di Biotecnologie a "La Sapienza" di Roma  , vorrei chiedere conferma, in virtù della presenza dei grandi esperti che frequentano questo forum: alla fine, quindi per verificare l'interazione tra un miRNA e un gene e il suo prodotto, quindi agendo in realtà sul suo mRNA, vado a fare due colture cellulari distinte di eucarioti quali piante o nematodi come C. elegans in cui i miRNA sono ben caratterizzati. , vorrei chiedere conferma, in virtù della presenza dei grandi esperti che frequentano questo forum: alla fine, quindi per verificare l'interazione tra un miRNA e un gene e il suo prodotto, quindi agendo in realtà sul suo mRNA, vado a fare due colture cellulari distinte di eucarioti quali piante o nematodi come C. elegans in cui i miRNA sono ben caratterizzati.
-In una linea cellulare faccio overesprimere, transfettando con un vettore, un miRNA specifico per un mRNA con prodotto proteico noto in modo da inibire o limitare la sua produzione mentre lascio intatta l'altra linea, usandola come controllo.
-Faccio il Western blot per verificare i diversi livelli di produzione proteica, dovrei vedere che nella linea cellulare transfettata c'è assenza o quantità limitata della proteina rispetto alla linea cellulare di controllo.
Quindi in realtà è molto semplice vista così no? Anche perchè credo che le regione 3'-UTR siano più complicate da modificare e forse meno attendibili per certi aspetti (almeno secondo la mia impressione). Crreggetemi se ho detto una castroneria! 
PS: il suddetto professore una volta ho posto un'altra domanda dello stesso stampo: come si potrebbe dimostrare che il dominio CTD della RNA Polimerasi II influisce sui processi di splicing e poliadenilazione? A quanto ho capito non conviene cercare di modificare il suddetto dominio CTD (a causa della difficoltà e di reazioni troppo forti come ad esempio il fatto che nemmeno partirebbe la trascrizione) ma piuttosto andare a influenzare quelle molecole come i TF e specialmente con il capping enzyme, che potrei marcare con anticorpo e poi immunoprecipitare, è corretta come procedura? Grazie mille comunque a tutti!
PS2:per il lievito S.pombe ci sono studi, tanto che la V edizione italiana del Watson ne parla, ma riguardano il silenziamento genico mediato dalle modificazioni della cromatina nelle regioni centromeriche e riguardano i siRNA (short interfering RNA). |
 |
|
|
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|





 ...
...
