| Autore |
Discussione |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 25 agosto 2009 : 12:38:45 Inserito il - 25 agosto 2009 : 12:38:45




|
Salve ragazzi, chi se ne intende di PCR?
Non riesco a capire perchè nel seguente sistema di mutagenesi sito-diretta, i due primers, riconoscendo l'uno l'elica positiva e l'altro l'elica negativa del DNA bersaglio non si appaino tra di loro. Qualcuno potrebbe essere così gentile da spiegarmelo? Penso c'entri qualcosa la temperatura di melting dei primers
Immagine:
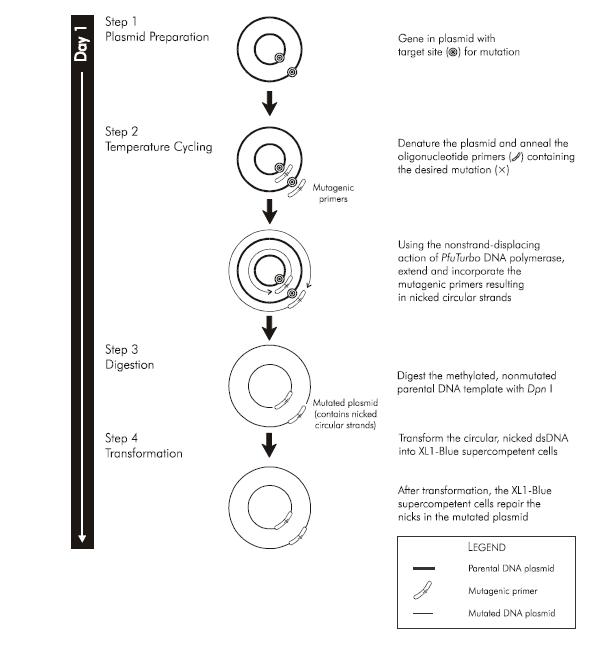
42,68 KB.
GRAZIE!!!
|
|
|
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 25 agosto 2009 : 13:45:51 Inserito il - 25 agosto 2009 : 13:45:51




|
Per curiosità sono anche andato sul sito della ditta [http://www.stratagene.com/sdmdesigner/default.aspx]che produce il kit ed ho progettato due primer per aggiungere due nucleotidi(A e T per la precisione) tra i nucleotidi 89 e 90 di questa sequenza:
ttttcagtcatcaataacatttttatttcaggtacagctgcaactaggtagaacaatgaattttgttagactagttccatttcagggaggcgtcaaatttatggctgggcctgtcatagcaatttttaacaaaatcatga
actcaaatctgtagcaacttttaaagatgtacttatttccaaatcagattacaacctaaggttacaacag
tccagactgcatccatttttttccagtagtagaaagaatgtatgataaactcattctacaaacaccactcaggaactctgcatcactaaggtcactttaaattgcactgtttacaaattacaccctttaccaaaaacgcagcaa
Il risultato come si può notare sono due primers perfettamente complementari.Ora,chi mi dice che mettendo su una PCR sti due primers non annilino fra di loro anzicchè al mio inserto, come rappresentato sopra?
|
 |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 25 agosto 2009 : 13:51:04 Inserito il - 25 agosto 2009 : 13:51:04




|
Immagine:
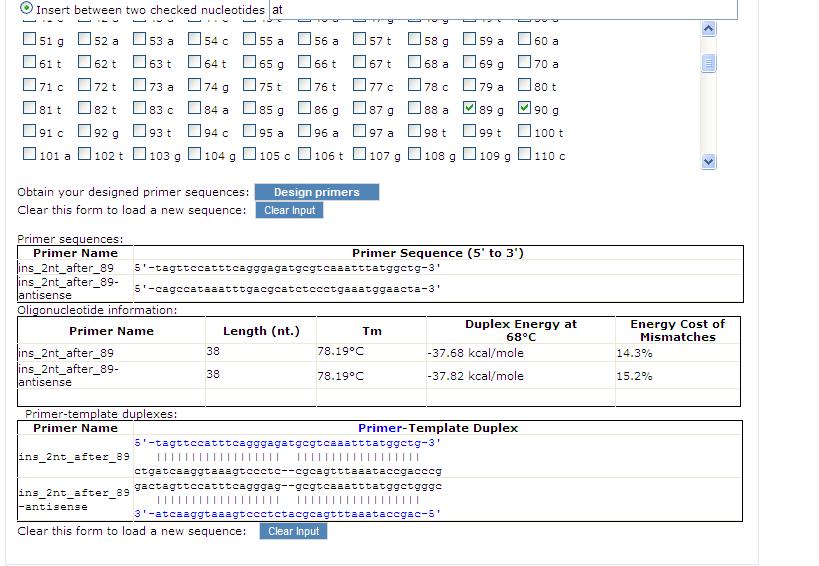
92,37 KB |
 |
|
|
domi84
Moderatore
  

Città: Glasgow

1724 Messaggi |
 Inserito il - 25 agosto 2009 : 14:21:12 Inserito il - 25 agosto 2009 : 14:21:12




|
Indubbiamente i primer si appaieranno anche fra di loro, ma in quel caso non avviene nessuna amplificazione, perchè la polimerasi non avrà nulla da polimerizzare:
5'------------3'
3'------------5'
Nel caso del primer invece che annila sul plasmide c'è da polimerizzare e quindi amplificazione... |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
tambua
Nuovo Arrivato

50 Messaggi |
 Inserito il - 25 agosto 2009 : 15:08:17 Inserito il - 25 agosto 2009 : 15:08:17




|
Buono a sapersi dottore :)
Grazie |
 |
|
|
Frin74
Nuovo Arrivato

17 Messaggi |
 Inserito il - 09 aprile 2010 : 01:16:29 Inserito il - 09 aprile 2010 : 01:16:29




|
Salve avrei bisogno di un'informazione, ovvero, dovrei fare una mutagenesi sito diretta cambiando solo UNA base in un plasmide. Nel mio lab abbiamo gia' il kit della stratagene per la MULTI mutagenesi sito diretta, ovviamente posso usare questo kit senza dover comprare quello per la singola mutagenesi, ma mi chiedo quanti primer devo utilizzare (perche' ovviamnte se ne usano tanti quante mutazioni devi introdurre e nel mio caso e' solo una)...sembrerebbe che io ne debba usare uno solo, ma non si capisce! chi ha esperienza in questo campo e soprattutto con questo caso specifico?
grazie
Frin |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 09 aprile 2010 : 01:37:13 Inserito il - 09 aprile 2010 : 01:37:13





|
scusa,ma tu la mutazione la metti sul primers e poi fai il resto ,non capisco il problema ,
anzi mi spiegheresti la differenza tra il kit per le multimugenesi e quello della singola? |

|
 |
|
|
Frin74
Nuovo Arrivato

17 Messaggi |
 Inserito il - 09 aprile 2010 : 18:31:37 Inserito il - 09 aprile 2010 : 18:31:37




|
I gotta
se uso solo un primer otterro' un solo strand (dopo la digestione con enzima DpnI) ma non e' un problema perche' poi le cellule ultracompetenti del kit sono adattate per essere trasformate con ssDNA e poi sintetizzano la'altro strand. |
 |
|
| |
Discussione |
|





