| Autore |
Discussione |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 19 novembre 2010 : 10:50:23 Inserito il - 19 novembre 2010 : 10:50:23




|
Ciao a tutti, volevo avere qualche consiglio pratico sull'utilizzo del Kit "QuikChange site-directed mutagenesis" della Stratagene.
Devo introdurre 3 mutazioni in una sequenza di 1800bp contenuta in un vettore di circa 4000bp e mi chiedevo se è possibile farlo con un singolo primer. Le tre mutazioni da introdurre sono piuttosto vicine:
tra la prima e la seconda tripletta da mutare ci sono 21bp, mentre la seconda e la terza sono consecutive.
HELP ME!
Grazie per l'attenzione
|
|
|
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 19 novembre 2010 : 11:28:58 Inserito il - 19 novembre 2010 : 11:28:58





|
| Certo che si puo' fare, e' molto efficace , consireda che si possono introdurre inserzioni o delezioni, quindi vai tranquilla, immagino che tu volessi dire con una singola coppia di primers giusto? |

|
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 19 novembre 2010 : 12:06:17 Inserito il - 19 novembre 2010 : 12:06:17




|
E qui il punto...intendevo proprio con un primer solo perchè mi hanno assicurato nel laboratorio dove sono che in passato lo hanno fatto con un'unico primer (quindi appaiato ad un solo filamento)....ma io ho provato già 3 volte: dopo la trasformazione dei batteri con il prodotto di mutagenesi mi vengono le colonie, estraggo il DNA, lo faccio sequenziare, ma alla fine non ho ancora trovato un clone con le mutazioni desiderate...
Posso chiederti, se non sono inopportuna, cosa suggeriresti di fare?
Ti ringrazio per la tempestiva risposta
Saluti
Silvia |
 |
|
|
metionina
Nuovo Arrivato
Città: Cambridge

45 Messaggi |
 Inserito il - 19 novembre 2010 : 12:19:24 Inserito il - 19 novembre 2010 : 12:19:24




|
| un solo primer appaiato su un solo filamento?? ma ci riuscivano davvero?? io ho sempre usato una coppia! |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 19 novembre 2010 : 14:57:03 Inserito il - 19 novembre 2010 : 14:57:03





|
Immagine:
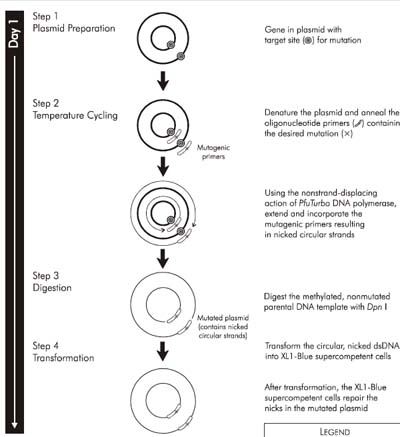
65,68 KB
Io direi che in questo modo abbassi di molto l'efficienza, tuttavia perche' cambiare il protocollo se e' scritto che bisogna farlo con 2 primers :) ? digerisci con DpN1 vero prima della trasforamzione , io ti suggerirei di prendere l'altro primers e seguire pari passo il protocollo , le mutazione cerca di metterle al centro ,non e' che ci sia molto da poter modificare |

|
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 19 novembre 2010 : 15:55:19 Inserito il - 19 novembre 2010 : 15:55:19




|
Ti ringrazio; effettivamente nel manuale di istruzione del kit che usiamo sono indicati 2 primer, ma sullo stesso filamento....ora non ho lo scanner, ma in questi giorni vedo di allegartelo... anch'io ero un po' perplessa, non avendone mai sentito parlare, ma riguardando il manuale d'istruzione sembra proprio così. Comunque sì, digerisco con Dpn1 prima di trasformare.
Altro problema, che persisterebbe anche seguendo il protocollo standard è che per avere le mutazioni al centro ho un primer di 60 nucleotidi...non è un po' troppo lungo?? |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 19 novembre 2010 : 18:24:13 Inserito il - 19 novembre 2010 : 18:24:13





|
vedi sul datasheet, magari dopo gli do un occhiata, tuttavia se vedi l'immagine che ti ho postato i primers sono appaiati sui 2 filamenti del vettore, anche perche' e' quello che usi come template, come faresti a farlo a singolo filamento , non ti meravigliare dei primers se sono lunghi, sono fatti apposta per far si che ci sia l appaiamento anche se ci sono missmatch, alla fine cio' che interessa e' che cia un corretto appaiamento nella regione 3' del primer , non ti meravigliare dei primers se sono lunghi, sono fatti apposta per far si che ci sia l appaiamento anche se ci sono missmatch, alla fine cio' che interessa e' che cia un corretto appaiamento nella regione 3' del primer |

|
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 22 novembre 2010 : 01:50:27 Inserito il - 22 novembre 2010 : 01:50:27




|
Eccomi, non mi sono dimenticata. Ho messo in allegato il datasheet che ho usato (che è diverso da quello che mi hai mandato tu). I primer in questo caso sono 2, ma indicati sullo stesso filamento nella stessa direzione...e il filamento complementare viene prodotto in vivo dai batteri immagino... Cosa ne pensi? Ho interpretato male io? Tuttavia io ho modificato ulteriormente questo datasheet utilizzando un solo primer (più lungo di quanto suggerito)...
Immagine:

481,1 KB |
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 22 novembre 2010 : 01:51:56 Inserito il - 22 novembre 2010 : 01:51:56




|
| Scusami l'hi inserita enorme l'immagine...era la prima volta che allegavo un file sul forum...sorry, farò più attenzion la prossima volta |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 22 novembre 2010 : 12:06:56 Inserito il - 22 novembre 2010 : 12:06:56





|
Non ti preoccupare
Allora, sinceramente questo kit sara' la versione precedente di quello che ho usato io, tuttavia avra' sicuramente un efficienza piu' bassa rispetto all'ultimo che hanno fatto, io ti suggerirei una cosa, essendo che alla fine cio' che conta di questo kit e' l'enzima Dpn1, ordina l'altro primer con la mutazione nella stessa posizione del tuo forward, per la reazione di PCR credo che il protocollo sia uguale, usano entrambi una Pfu e per la temperatura di anniling puoi usare la stessa equazione riportata nel datasheet, poi segui come dice il protocollo, digerisci e trasformi
fammi sapere, non saprei dirti piu' di questo |

|
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 23 novembre 2010 : 15:20:50 Inserito il - 23 novembre 2010 : 15:20:50




|
Il datasheet che ho io dà un'efficienza del 100%...però rispetto a ciò che è indicato noi abbiamo modificato un po' di cosette:
un solo primer anzichè 2
LB anzichè NZY+ (terreno usato durante la trasformazione)
Per il resto abbiamo seguito il protocollo indicato, ma può darsi che il primer fosse troppo lungo o che insierire 3 mutazioni contemporaneamente abbassi notevolmente l'efficienza.
Per il momento ho accantonato la cosa, ma la prossima settimana riinizio. Ti farò sapere, grazie. |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 23 novembre 2010 : 20:27:04 Inserito il - 23 novembre 2010 : 20:27:04





|
| Addirittura del 100% ma che bugiardi :D |

|
 |
|
|
agny84
Nuovo Arrivato

1 Messaggi |
 Inserito il - 23 novembre 2010 : 21:00:57 Inserito il - 23 novembre 2010 : 21:00:57




|
Ciao a tutti, ho provato ad utilizzare il QuikChange site-directed mutagenesis della stratagene due volte ma ho avuti un po di problemi. Devo inserire due mutazioni puntiformi in punti molto vicini in un inserto di 3.2 Kb in un plasmide di 3 Kb.
Ho fatto la PCR seguendo il protocollo e impostandola a 12 cicli per le mutazioni puntiformi. Ho verificato la PCR e le bande le ho viste anche se nel protocollo c'e' scritto che potrebbero anche non vedersi e successivamente ho proseguito con la digestione con Dpn1, e fin qui apparentemente ok.
Procedo con la trasformazione e l'unica cosa che non seguo come da protocollo e' l'aggiunta dell'x-gal e dell'IPTG nelle piastre con l'antibiotico perche' il mio plasmide non richiede lo screening con l'x-gal.
Le colonie crescono(con un aspetto non bellissimo quasi lattescente), estraggo il Dna dalle colonie,ovviamente dopo essere state messe in coltura o/n,faccio la digestione con un enzima che taglia il mio inserto dal plasmide ,per verificare se effettivamente e' avvenuta la trasformazione, e vedo che le colonie sono vuote.
Cosa succede secondo voi???
help me,please
|
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 26 novembre 2010 : 16:53:13 Inserito il - 26 novembre 2010 : 16:53:13




|
Ciao, rispondendo a Patrizio: e già, sono proprio dei bugiardi, guardando i miei risultati direi che l'efficienza è dello 0% visto che nei pochi casi in cui ho ottenuto delle mutazioni non erano comunque quelle desiderate!! (In alcuni casi ho riscontrato frame-shift e inserzioni...). Vabbè, ora non ci voglio più pensare fino a lunedì...Ti aggiornerò quando neverrò a capo :D
Rispondendo ad Agny84: Figurati che io di mutazioni puntiformi ne devo inserire 3!! A parte tutto...Spero di esseri di aiuto, ma premetto che non sono un'esperta:
Secondo il protocollo che ho io sono da fare 30 cicli di PCR
Il tuo inserto è più grande del plasmide?
Comunque se le colonie crescono in un terreno con antibiotico le cose (per il poco che ne so) possono essere 2:
- o la trasformazione è avvenuta( e in questo caso però non è detto che il vettore sia ricombinante, ovvero non è detto che ci sia l'inserto)
oppure i batteri sono resistenti all'antibiotico... dovresti fare un controllo piastrando batteri non trasformati, se ti vengono comunque le colonie i batteri sono di per se stessi resistenti e non puoi usarli.
 |
 |
|
|
silvietta82
Utente Junior


126 Messaggi |
 Inserito il - 03 dicembre 2010 : 12:17:58 Inserito il - 03 dicembre 2010 : 12:17:58




|
Ciao a tutti di nuovo io!!! Ho disegnato nuovi primer...appena li ho e ho cellule competenti ricomincio!
Mi è sorto un ulteriore dubbio: il mio templato proviene da E. Coli TOP F1-10.
Qualcuno mi sa dire se questo ceppo è dam+ ( oppure sapete dirmi dove trovare questa informaziione) ??
Perchè se il ceppo fosse dam- l'enzima Dpn1 non digerisce il parentale.... chiedo ancora questo aiuto, spero sia risolutivo
Ciao a tutti e grazie mille :) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
|
| |
Discussione |
|






 , non ti meravigliare dei primers se sono lunghi, sono fatti apposta per far si che ci sia l appaiamento anche se ci sono missmatch, alla fine cio' che interessa e' che cia un corretto appaiamento nella regione 3' del primer
, non ti meravigliare dei primers se sono lunghi, sono fatti apposta per far si che ci sia l appaiamento anche se ci sono missmatch, alla fine cio' che interessa e' che cia un corretto appaiamento nella regione 3' del primer

