| Autore |
Discussione |
|
|
totka
Nuovo Arrivato

6 Messaggi |
 Inserito il - 30 maggio 2012 : 13:25:28 Inserito il - 30 maggio 2012 : 13:25:28




|
ciao a tutti!!
ho un problema con un gel di agarosio 0,8% fatto per verificare la presenza di un amplificazione in pcr( sto cercando di fare una mutazione sito diretta)
dopo la pcr digterisco il dna templato con dpn1 per 30minuti a 37°C.
nel pozzetto metto 1,5µ di cybrgreen e 5µl di campione dalla PCR, dopo averli mescolati e tenuti in ghiaccio per 30minuti.
il gel corre a 100V per 1ora.
il marker migra una meraviglia, ma i miei campioni no!
ho provato a far correre anche il templato nudo e crudo dopo
l´estrazione e questo migra perfettamente!
dalla foto del gel con il transilluminatore si vede chiaramente che i pozzetti dopo un´ora sono ancora pieni.
qualcuno ha un idea??  
|
|
|
|
|
roberta.s
Utente Junior


Città: Parigi

564 Messaggi |
 Inserito il - 30 maggio 2012 : 13:53:23 Inserito il - 30 maggio 2012 : 13:53:23




|
non ho capito il tuo esperimento. ad ogni modo, hai fatto un'elettroforesi dopo PCR (prima della digestione) per verificare che quest'ultima abbia funzionato? hai un controllo positivo?
poi, da NEB leggo che anche DpnI va utilizzato almeno 1h a 37°C. Questo non c'entra con il tuo problema, ma credo che mezz'ora sia un po' poco. |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 30 maggio 2012 : 21:59:35 Inserito il - 30 maggio 2012 : 21:59:35




|
| nemmeno a me è chiaro esattamente cosa hai fatto. Potresti indicare cosa hai caricato nei pozzetti? |
 |
 |
|
|
totka
Nuovo Arrivato

6 Messaggi |
 Inserito il - 31 maggio 2012 : 10:12:42 Inserito il - 31 maggio 2012 : 10:12:42




|
cercheró di essere piú chiaro:
sto cercando di fare introdurre una mutazione sito diretta in un gene precedentemente clonato all´interno del plasmide pET28a. per fare ció sto usando la PCR, quindi dopo questa faccio un gel pdi agarosio allo 0,8% per vedere se c´é stata un´amplificazione.
subito dopo la PCR digerisco il DNA templato con DpnI, poi prelevo 5µl di campione e li metto in una nuova E.cup con 1,5 µl di SYBRgreen, mescolo con la pipetta e tengo in ghiaccio per 20-30minuti.
nei pozzetti metto il mio campione mescolato con il SYBRgreen. dopo 1 ora a 100V il marker é migrato perfettamente, il plasmide estratto anche, ma i campioni della PCR non si muovono dai pozzetti.
sono stato abbastanza chiaro??
  |
 |
|
|
SpemannOrganizer
Utente
 

Città: Los Angeles

955 Messaggi |
 Inserito il - 31 maggio 2012 : 11:00:51 Inserito il - 31 maggio 2012 : 11:00:51




|
| Perchè usi la pcr e non il kit per introdurre la mutazione? Ad ogni modo, sei sicuro che la tua pcr funzioni? nel pozzetto della pcr, il plasmide dovrebbe essere degradato completamente perchè digerito da DpnI (a meno che nn sia stato metilato, questo dipende dal ceppo di batteri che usi), quindi dovresti vedere solo la banda della pcr. A che altezza dovrebbe migrare l'amplificato? Dove si appaiano i primers? che regione dovrebbero amplificare? |
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 31 maggio 2012 : 11:19:40 Inserito il - 31 maggio 2012 : 11:19:40





|
Beh con il kit sarebbe uguale, fai una PCR con i primers mutageni e poi digerisci con DpnI!
totka hai provato a seminare anche l'amplificato senza averlo digerito con DpnI? Potrebbe essere un buon controllo.
Sinceramente non mi sembra poi così strano che tu non veda migrazione!
Tieni presente che il plasmide mutagenizzato che ottieni dopo PCR è circolare con un doppio nick, questa è la forma "più pesante" e migrerà quindi molto più lentamente rispetto alle altre forme (ad es. il superavvolto che vedi nel plasmide di partenza).
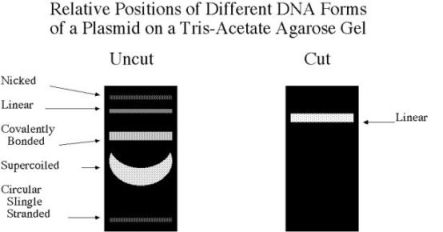
Probabilmente devi far migrare molto più a lungo per poter vedere una migrazione.
Mi spiace ma una tempistica esatta dei tempi di migrazione non te la so dare perché praticamente non l'ho mai fatto, ho sempre fatto migrare plasmidi circolari o lineari. |
 |
|
|
totka
Nuovo Arrivato

6 Messaggi |
 Inserito il - 31 maggio 2012 : 11:37:56 Inserito il - 31 maggio 2012 : 11:37:56




|
grazie GFPina!! questa non la sapevo o comunque non la ricordavo, ieri ho provato a trasformare comunque i miei campioni e oggi ho qualche colonia, domani mando a sequenziare e incrocio le dita!    |
 |
|
|
totka
Nuovo Arrivato

6 Messaggi |
 Inserito il - 31 maggio 2012 : 17:23:08 Inserito il - 31 maggio 2012 : 17:23:08




|
allora, questa mattina ho fatto un gel dopo pcr senza digerire con DpnI, l´unica banda che mi compare é l´ultima(75b), poi nel pomeriggio ne ho fatto un´altro dopo digestione con DpnI, ma la banda si é spostata a 200b.la cosa pi´´u problematica é che mi aspettavo di vedere il mio templato (7kb) senza digerire con DpnI. devo pensare di avere una contaminazione da nucleasi? inizio a non capirci piú niente...  |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 01 giugno 2012 : 22:48:24 Inserito il - 01 giugno 2012 : 22:48:24





|
Scusa ma non mi è molto chiaro, potresti spiegare meglio?
Comunque se hai ottenuto delle colonie magari è probabile che siano quelle mutagenizzate, aspettiamo i risultati delle sequenze!  |
 |
|
|
Aul_ICOS
Nuovo Arrivato

25 Messaggi |
 Inserito il - 01 giugno 2012 : 23:41:32 Inserito il - 01 giugno 2012 : 23:41:32




|
hai provato a farlo correre meno?
|
 |
|
| |
Discussione |
|








