| Autore |
Discussione |
|
|
Lisa.ody
Nuovo Arrivato

43 Messaggi |
 Inserito il - 29 gennaio 2008 : 21:53:40 Inserito il - 29 gennaio 2008 : 21:53:40




|
|
per trovare un gene ke dovrei fare? mi potete kiarire come si fa lo screening di librerie genomike e mi potreste spiegare tutte le tecnike ke posso usare per identificare un gene?
|
|
|
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 29 gennaio 2008 : 22:16:18 Inserito il - 29 gennaio 2008 : 22:16:18





|
| dipende da cosa parti...cos hai a disposizione |

|
 |
|
|
Lisa.ody
Nuovo Arrivato

43 Messaggi |
 Inserito il - 29 gennaio 2008 : 22:40:51 Inserito il - 29 gennaio 2008 : 22:40:51




|
| vorrei avere un quadro generale di tutte le poss situazioni...anke in sintesi poi magari vedo d approfondire da sola... |
 |
|
|
Nick_row
Utente Junior



361 Messaggi |
 Inserito il - 29 gennaio 2008 : 22:43:55 Inserito il - 29 gennaio 2008 : 22:43:55




|
| per trovare un gene...è un pò generico:il gene è noto?è stato mappato e clonato?allora fish!non è noto,non è stato mappato?allora linkage... |
 |
|
|
abby
Nuovo Arrivato

Città: viterbo

17 Messaggi |
 Inserito il - 29 gennaio 2008 : 22:46:51 Inserito il - 29 gennaio 2008 : 22:46:51




|
lo screening delle genoteche si basa su 2 principi a seconda dei casi:
screening per sequenza e
screening per funzione,che si applica se non si ha nessuna informazione a livello di sequenza e posizione del gene.
per identifgicare una sequenza clonata si usano varie tecniche tipo l'ibridazione con sonde radioattive o primer sintetizzati con pcr.
per identificare un gene di cui non si sa la seq e non si possono costruire sonde si studia il prodotto genico,quindi si individuano le proteine con vari metodi,uno è il saggio enzimatico...
tutte le tecniche si trovano sul primrose...spero di essere stata utile |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 29 gennaio 2008 : 23:08:03 Inserito il - 29 gennaio 2008 : 23:08:03





|
Immagine:
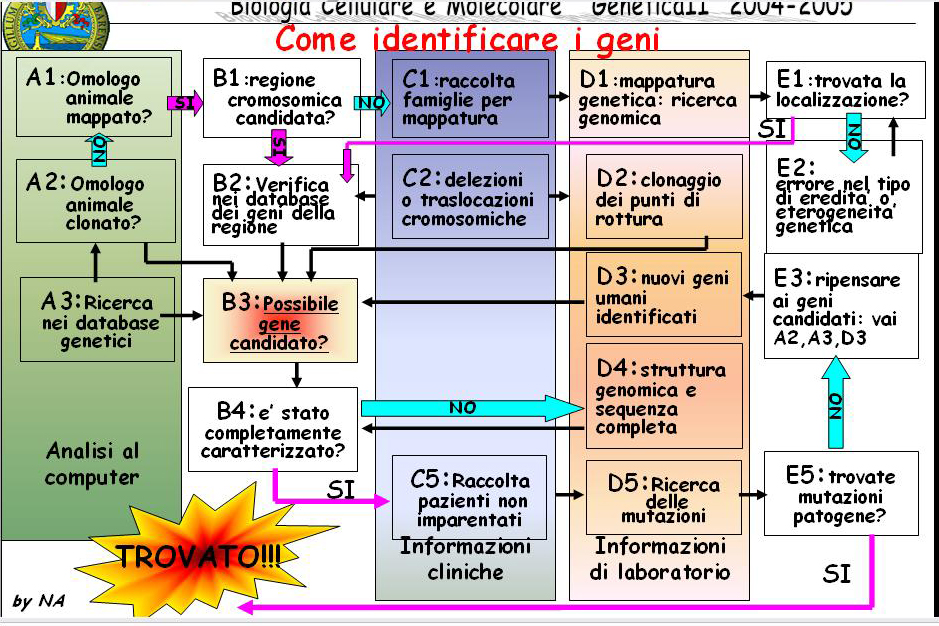
555,56 KB
trova il tuo punto |

|
 |
|
|
Lisa.ody
Nuovo Arrivato

43 Messaggi |
 Inserito il - 29 gennaio 2008 : 23:26:57 Inserito il - 29 gennaio 2008 : 23:26:57




|
mamma mia..nn riesco a seguirlo + d tanto come skema...
allora io so come s fa un clonaggio..si ottengono banke genomike, cromosomiche o d cDna...dopo di ke per trov un sts o un gene è necessario uno screening o radioattivo o mediante pcr ma in ke cosa consistono precisamente? lo screening d queste librerie si può fare quando il gene si conosce?
quando s conosce solo la funzione devo utilizzare solo il saggio di una banca di cDna?
e il linkage in che categoria lo metto? e come si usa? |
 |
|
|
Lisa.ody
Nuovo Arrivato

43 Messaggi |
 Inserito il - 30 gennaio 2008 : 11:15:46 Inserito il - 30 gennaio 2008 : 11:15:46




|
| vi prego rispondete...domani ho l'esame...:| |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 30 gennaio 2008 : 19:44:51 Inserito il - 30 gennaio 2008 : 19:44:51





|
I mezzi come ti ho scritto possono variare,le scelte dipendono dalle informazioni di partenza...che poi possono integrarsi durante le tue ricerche( praticamente alla fine possono anche usarsi tutte le tecniche che conosci!! ),non esiste quella giusta in assoluto devi trovare quella che in base a cio' che hai ti da maggiori risultati..e da li poi andare avanti e "tassello per tassello" lo becchi
Bene..se sai tutto questo,vediamo se mettiamo in ordine....innanzitutto devi sapere ovviamente la funzione o il carattere che il gene ti da ovviamente,oppure come in questo caso il prodotto del gene....ma non sai dove si trova! ,in questo caso puoi costruirti degli ibridi somatici( in genere si fondono cellule di topo con quelle di uomo ) ,in questo caso puoi costruirti degli ibridi somatici( in genere si fondono cellule di topo con quelle di uomo )
Una volta trovato il cromosoma...ti fai le mappe con ibridi da radiazione e ti ricavi anche la regione subcromosomica in cui mappa il tuo gene!....ripeto,questo quando tu conosci il prodotto ma non il gene!
Nel caso tu voglia trovare ...tipo un gene malattia,non conoscendo ne il prodotto ne la localizzazione,la strada e' un po' piu' lunga...innanzitutto devi trovare un polimorfismo che si trova in linkage visto che non sai niente del gene, ma sai solo che il locus e' da qualche parte nel genoma e quando mutato rompe! ,(inizialmente si puo’ usare anche un polimorfismo proteico,se sai dove mappa e’ bene altrimenti saprai solo che e’ in linkage con questo) ,(inizialmente si puo’ usare anche un polimorfismo proteico,se sai dove mappa e’ bene altrimenti saprai solo che e’ in linkage con questo)
quindi vai a testare tutte le famiglie in cui e’ presente almeno un affetto con polimorfismi del DNA di cui si conosce la localizzazione,quando hai trovato il linkage HAI MAPPATO FISICAMENTE IL LOCUS!!
….pero’ non hai trovato il gene;..pero’ sai dove andarlo a cercare 
Per una patologia autosomica recessiva l analisi di linkage e’ ostacolata dal fatto che non si conosce la fase,quindi lo considere prima in cis e poi in trans e il lod score sara’ la media dei 2 valori ottenuti,a questo punto pero’ ,devi andare a vedere se ci sono altri marcatori e vedere se sono i responsabili,se sono ricombinanti…non saranno loro il nostro gene d interesse,pero’ il punto e’ cercarne quanti piu’ puoi,in maniera tale da restringere la regione d interesse… nel cado del gene della CF,fu usato anche il cromosome jumping…. ,ora vedi se c’e’ linkage disequilibrium con un marcatore,ovviamente anche se hai trovato l allele associato…non e’ detto che sempre sia associato con il gene d interesse!!!ma se la percentuale di quell aplotimo e’ altissima come in genere accade non ci sono dubbi
A questo punto mediante zoo-blot,gene walking e cromosome jumping puoi clonarti il gene
se non ti e' chiara qualcosa chiedi pure
...in bocca al lupo per domani |

|
 |
|
|
Lisa.ody
Nuovo Arrivato

43 Messaggi |
 Inserito il - 30 gennaio 2008 : 21:13:07 Inserito il - 30 gennaio 2008 : 21:13:07




|
oook grazie patrizio!!
nn ho ben capito come faccio a ricavarmi il cromosoma tramite gli ibridi da radiazione. dunque io so ke se con gli ibridi da radiazione si costituisce una nuova linea ibrida in cui però nn tutti i cromosmi vengono integrati all'interno del nucleo e quindi è necessaria una caratterizzazione utilizzando il sistema ALU-PCR seguito da una Fish e così ottengo pannelli di ibridi, ma dopo ke ho ricavato questi pannelli ke dovrei fare?come s procede?
il prof c parlò anke di una caratterizzazione + fine mediante STS e PCR per ottenere pannelli di singoli cromosomi e del souther genomico di un ibrido per analizzare nello specifico un gene, cn il risultato ke il gene ke cerkiamo deve ess sempre presente negli ibridi positivi e sempre assente in quelli negativi. |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 30 gennaio 2008 : 22:29:06 Inserito il - 30 gennaio 2008 : 22:29:06





|
Questa tecnica si basa sul fatto che cellule sottoposte a radiazioni letali(RX) vengono recuperate per mezzo di una fusione con adatte cellule riceventi, e' piu' utile partire da ibridi contenenti un solo cromosoma umano che vengono irradiati e poi fusi. La tappa di irradiazione ha due funzioni fondamentali.
Primo, la dose letale di radiazione è necessaria per assicurare la morte di tutte le cellule donatrici, generalmente dosi maggiori di 1500 rad sono sufficienti per uccidere la maggior parte dei vari tipi di cellule.
Secondo, l'irradiazione causa rotture a livello del DNA e quindi determina la frammentazione dei cromosomi; aumentando la dose di radiazione aumenta la frequenza di rotture cromosomiche.
Dopo l'irradiazione, le cellule donatrici vengono fuse con cellule di topo riceventi utilizzando il PEG.
La linea cellulare ricevente deve essere deficiente per qualche marker che permetta poi la selezione a favore dell'ibrido.
Si possono utilizzare cellule riceventi che siano HGPRT- e quindi in tal caso si fa uso di HAT per la selezione.
Molto spesso il cromosoma umano di partenza viene reso resistente all'antibiotico G418 mediante integrazione del gene neo in diverse copie del cromosoma umano, per cui le cellule che possiedono materiale umano sono resistenti al G418.
Gli ibridi che si ottengono contengono in genere frammenti umani inseriti nei cromosomi di roditore. Il DNA estratto da questi ibridi viene amplificato tramite una Alu-PCR (le sequenze Alu si trovano solo nel DNA umano ogni 4-5Kb circa); i prodotti sono quindi utilizzati come sonde in esperimenti di FISH su metafasi umane normali per un accurata caratterizzazione citogenetica di materiale umano ritenuto in ogni ibrido
Detto questo....serve per mappare ad esempio una regione subcromosomica che abba subito delezione o traslocazione( non il gene ),cioe' tu sai che i tuoi ibridi..hanno un cromosoma aberrante,quindi mediante le radiazioni frammenti quel cromosoma fai fondere con una cellula di topo,alcuni di questi si integrano e avrai tanti ibridi con diversi frammenti di quel cromosoma ok?...quindi...ora mediante la ALU-PCR vai a ricavarti le sonde...le ALU sono molto presenti nel genoma ,con queste sonde di ciascun ibrido, vai a fare FISH su cromosomi metafasici e avrai questo
Immagine:
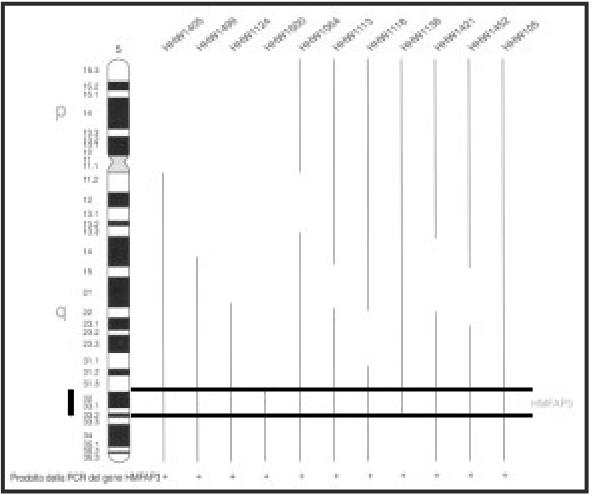
27,57 KB (le barre verticali sono le estenzioni del cromosoma)
a questo punto,se vedi che una tua sonda mappa su un cromosoma metafasico diverso dal primo,vuol dire che li e' il punto in cui e' avventuta la traslocazione! |

|
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 31 gennaio 2008 : 00:56:03 Inserito il - 31 gennaio 2008 : 00:56:03





|
| Ormai e' tardi....ma cmq...non me lo ricordo come cacchio si usano gli ibridi da radiazione per identificare la banda o il locus..e in rete non ho trovato niente!!!..cmq posso pensare che praticamnente uso l ibrido che e' mutato per quel gene...costringendolo ad avere quello nel cromosoma umano...(usando anche G418),e poi anche qui ti fai le sonde con ALU-PCR e le FISH...con questo ragionamento mi torna..ma e' una cosa mia..sballata |

|
 |
|
| |
Discussione |
|






