| Autore |
Discussione |
|
|
stef2
Nuovo Arrivato

21 Messaggi |
 Inserito il - 09 maggio 2008 : 10:47:36 Inserito il - 09 maggio 2008 : 10:47:36




|
Ciao, ho diversi grandi dilemmi (AIUTATEMI VI PREGO!!):
1)Qualcuno potrebbe inviarmi il protocollo per preparare DNA o RNA per la lettura allo spettrofotometro?(Es. come bianco cosa si usa?)
2)Qualcuno ha un protocollo per analizzare la metiliazione di alcuni geni cioè le famose isole cpg nei promotori dei geni?
3)Devo calcolare per la prima volta la concentrazione dei primers(cioè li ho liofilizzati e li devo preparare in uno stock)per fare una pcr:come devo fare?
mi hanno dato una formula (presa dal libro Maniatis) che per me non è tanto comprensibile:
ES. un oligo lungo 20 bp= 154 picomoles di DNA in 1 gamma (che significa?)
se 154:1gamma=1:x
x=1/154=0.00649 pmol/1000
1 pic=6.5ng di DNA
5umol/L=5nmol/mL=5pmol/ul
per avere 5 pmol/uL si moltiplica 6.5 ng*5=32.5 ng/uL
NULLA MI E' CHIARO IN QUESTO CALCOLO!!!!
se ho un oligo più lungo di 20bp come faccio a calcolare la concentrazione?
154 picomoles perchè corrispondono a 20 bp?
e poi la proporzione 154:1gamma=1:x da dove deriva? e 1 nella proporzione da dove deriva?
PURTROPPO NON HO IL LIBRO ERANO FOTOCOPIE DI UNO STRALCIO DI APPUNTI...
4)Ho ancora una grande confusione sulle equivalenze per calcolare le concentrazioni:
1mM=100pmol?
100uM=100pmol/uL ? oppure
100uM=100umol/lt?
mi hanno dato queste equivalenze ma io credevo che 100uM=100umol/ul
mi date qualche dritta su come fare queste equivalenze/conversioni?
grazie a tutti
|
stef2 |
|
|
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 09 maggio 2008 : 15:57:02 Inserito il - 09 maggio 2008 : 15:57:02





|
Ciao! Vediamo di risolvere quelcuno dei tuoi dubbi! 
Citazione:
Messaggio inserito da stef2
1)Qualcuno potrebbe inviarmi il protocollo per preparare DNA o RNA per la lettura allo spettrofotometro?(Es. come bianco cosa si usa?)
Prova a guardare questa discussione in cui se ne è parlato:
http://www.molecularlab.it/forum/topic.asp?TOPIC_ID=3863
non è una cosa tanto difficile, ma se hai altri dubbi chiedi.
Come soluzione di lettura per l'RNA al posto dell'acqua è meglio utilizzare TE (10mM Tris-HCl pH8, 1mM EDTA).
Citazione:
2)Qualcuno ha un protocollo per analizzare la metiliazione di alcuni geni cioè le famose isole cpg nei promotori dei geni?
Questo non ce l'ho mi spiace! 
Citazione:
3)Devo calcolare per la prima volta la concentrazione dei primers(cioè li ho liofilizzati e li devo preparare in uno stock)per fare una pcr:come devo fare?
mi hanno dato una formula (presa dal libro Maniatis) che per me non è tanto comprensibile:
ES. un oligo lungo 20 bp= 154 picomoles di DNA in 1 gamma (che significa?)
se 154:1gamma=1:x
x=1/154=0.00649 pmol/1000
1 pic=6.5ng di DNA
5umol/L=5nmol/mL=5pmol/ul
per avere 5 pmol/uL si moltiplica 6.5 ng*5=32.5 ng/uL
NULLA MI E' CHIARO IN QUESTO CALCOLO!!!!
se ho un oligo più lungo di 20bp come faccio a calcolare la concentrazione?
154 picomoles perchè corrispondono a 20 bp?
e poi la proporzione 154:1gamma=1:x da dove deriva? e 1 nella proporzione da dove deriva?
PURTROPPO NON HO IL LIBRO ERANO FOTOCOPIE DI UNO STRALCIO DI APPUNTI...
Allora cerchiamo di fare chiarezza... probabilmente il valore di 154 picomoles è stato preso da una tabella perchè in genere i calcoli si fanno al contrario rispetto a quelli che hai indicatu tu!
Cioè quello che è noto è il peso di un nucleotide, quindi da questo ricavi il peso di un oligo moltiplicando per il numero di basi e poi puoi ricavare le picomoli.
Ti faccio l'esempio del tuo caso:
Il peso medio di un nucleotide = 324.5Da (a volte si usa approssimato a 330Da)
Da questo ricavi il peso del tuo oligo moltiplicando per il n° di basi:
20 x 324.5gr/mol = 6490 gr/mol
siccome poi a te interessa avere pmol/ug faccio una serie di trasformazioni (passo a passo per essere chiara) in modo da arrivare ad avere un valore esperesso in ug/pmol:
6490 gr/mol = 6.49 gr/mmol = 0.00649 gr/umol = 0.00649 mg/nmol = 0.00649 ug/pmol
a questo punto facendo l'inverso ottieni:
1/(0.00649 ug/pmol) = 154pmol/ug
che è il valore da cui sei partita.
Il calcolo per passare a una concentrazione 5 pmol/uL (=5uM) penso tu lo abbia capito.
Citazione:
4)Ho ancora una grande confusione sulle equivalenze per calcolare le concentrazioni:
1mM=100pmol?
100uM=100pmol/uL ? oppure
100uM=100umol/lt?
mi hanno dato queste equivalenze ma io credevo che 100uM=100umol/ul
mi date qualche dritta su come fare queste equivalenze/conversioni?
grazie a tutti
Allora le molarità (M) sono sempre rispetto al litro!!!
1 M = 1 mol/L
1 mM = 1 mmol/L
1 uM = 1 umol/L
1 nM = 1 nmol/L
1 pM = 1 pmol/L
però ovviamente puoi dividere o moltiplicare per 1000 e cambiano le unità di misura, quindi:
1 uM = 1 umol/L
dividendo sia sopra che sotto per 1000 passi:
- da umol a nmol
- da L a mL
quindi:
1 uM = 1 umol/L = 1 nmol/mL
dividendo ancora un ultima volta per 1000:
1 uM = 1 umol/mL = 1 nmol/uL = 1 pmol/uL
In genere la concentrazione dei primers di esprime in uM che sono pmol/uL
(per una tabella delle unità di misura guarda su wikipedia: http://it.wikipedia.org/wiki/Milli_%28prefisso%29)
Infine ti segnalo un link dalla Directory Scientifica dove trovi dei tools per fare i calcoli: http://www.molecularlab.it/directory_scientifica/index.asp?cat=115
  |
 |
|
|
stef2
Nuovo Arrivato

21 Messaggi |
 Inserito il - 12 maggio 2008 : 09:53:25 Inserito il - 12 maggio 2008 : 09:53:25




|
GRAZIE!!finalmente ho le idee chiare!!
Sai in lab è difficile che qualcuno ti spieghi qualcosa, danno molto spesso per scontato tutto, ma purtroppo all'università non spiegano un granchè...
a me piace tanto la ricerca ma avevo paura di non capirla però ora veramente il mio approccio è diverso con le tue chiarificazioni.
un'ultima domanda ma per calcolare la concentrazione degli oligo utilizzo in ogni caso sempre il tuo schema...!!?!!
grazie ancora    |
stef2 |
 |
|
|
stef2
Nuovo Arrivato

21 Messaggi |
 Inserito il - 12 maggio 2008 : 10:22:53 Inserito il - 12 maggio 2008 : 10:22:53




|
Scusami l' ultimo dubbio che mi è rimasto nel calcolo della concentrazione degli oligo è:
la proporzione 154:1gamma = 1:x da dove deriva?
e 1 nella proporzione da dove deriva?
è una costante fissa?
anche 1 gamma è fisso?
GRAZIE!!! |
stef2 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 12 maggio 2008 : 18:26:11 Inserito il - 12 maggio 2008 : 18:26:11





|
Di niente figurati! 
Citazione:
Messaggio inserito da stef2
un'ultima domanda ma per calcolare la concentrazione degli oligo utilizzo in ogni caso sempre il tuo schema...!!?!!
grazie ancora   
Si in genere si procede in quel modo, non puoi sapere a priori quante picomoli ci sono in un nanogrammo di oligo senza delle tabelle, devi calcolarlo.
Citazione:
Messaggio inserito da stef2
Scusami l' ultimo dubbio che mi è rimasto nel calcolo della concentrazione degli oligo è:
la proporzione 154:1gamma = 1:x da dove deriva?
e 1 nella proporzione da dove deriva?
è una costante fissa?
anche 1 gamma è fisso?
GRAZIE!!!
Non sono costanti ma semplicemmente tu sai che in 1 gamma (ug) di un oligo di 20basi ci sono 154pmol --> quindi 154pmol/ug
e ti ricavi quanti ng ci sono in una picomole.
La proporzione sarebbe quindi:
154 pmol : 1 ug = 1 pmol : X ug
da cui:
X ug = 1 pmol/154 pmol x 1ug = 0,00649 ug = 6,49 ng
Ah una precisazione, non mi ero accorta prima:
non è corretto dire che "un oligo è lungo 20 bp" perché è a singolo filamento, quindi non sono "paia di basi", bisognerebbe utilizzare "mer" o "nt" anche se spesso vengono comunque indicati con "bp".
  |
 |
|
|
stef2
Nuovo Arrivato

21 Messaggi |
 Inserito il - 13 maggio 2008 : 09:02:47 Inserito il - 13 maggio 2008 : 09:02:47




|
GRAZIE,TUTTO CHIARO ORA!!!!!!!!!!!!!    |
stef2 |
 |
|
|
stef2
Nuovo Arrivato

21 Messaggi |
 Inserito il - 15 maggio 2008 : 19:39:24 Inserito il - 15 maggio 2008 : 19:39:24




|
Ciao,
rileggendo attentamente la tua risposta mi rendevo conto che tu hai detto che ci sono delle tabelle dove poter calcolare quante picomoli ci sono in un nanogrammo di oligo (o altro),potresti indicarmi dove posso trovarle o se puoi inviarmele,così da velocizzarmi nei calcoli per determinare la concentrazione degli oligo?
Ma quando leggo il data sheet allegato agli oligo liofilizzati, dovrebbero essere già presenti questi valori della tabella di cui mi accennavi?
Sai il mio dubbio è: saprò calcolare bene la concentrazione degli oligo liofilizzati per poi diluirli in maniera corretta per una pcr? 
se hai qualke dritta potresti indicarmela?
grazie ciao   |
stef2 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 15 maggio 2008 : 21:23:03 Inserito il - 15 maggio 2008 : 21:23:03





|
Citazione:
Messaggio inserito da stef2
Ciao,
rileggendo attentamente la tua risposta mi rendevo conto che tu hai detto che ci sono delle tabelle dove poter calcolare quante picomoli ci sono in un nanogrammo di oligo (o altro),potresti indicarmi dove posso trovarle o se puoi inviarmele,così da velocizzarmi nei calcoli per determinare la concentrazione degli oligo?
Le tabelle non dove si trovino perché non le ho mai utilizzate, ma in realtà non è niente di difficile da calcolare, come ti ho già detto:
sapendo che:
Il peso medio di un nucleotide = 324.5Da = 324.5 ug/umol
basta utilizzare la seguente formula per calcolare quante umol ci sono in un ug:
1/(n°basi x 324.5) = umol/ug
o quante pmol ci sono in un ug:
basta utilizzare la seguente formula per calcolare quante umol ci sono in un ug:
10^6/(n°basi x 324.5) = pmol/ug
oppure molto più semplicemente utilizzare dei tool tipo questo (che trovi nella directory scientifica):
http://molbiol.edu.ru/eng/scripts/index.html
Selezioni ssDNA, inserisci il numero di basi (in kb): in questo caso 20 basi = 0,02kb
selezioni le unità di misura: ad. es 1 ug e pmol e clicchi su Calculate...
come vedi il risultato è 154pmol/ug 
(quello che avevamo calcolato!)

Citazione:
Messaggio inserito da stef2
Ma quando leggo il data sheet allegato agli oligo liofilizzati, dovrebbero essere già presenti questi valori della tabella di cui mi accennavi?
Questo è un esempio di data sheet che potresti avere allegato al tuo oligo:
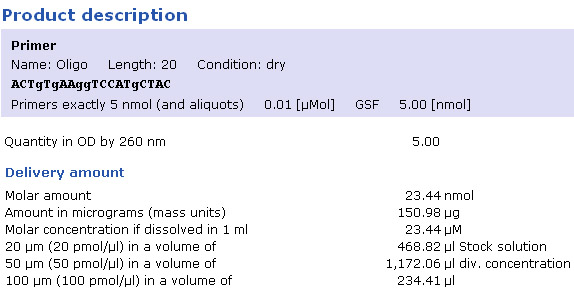
come vedi la maggior parte delle volte non devi neanche fare i conti, ti dicono già in quanto risospenderlo per avere la concentrazione desiderata!
Citazione:
Sai il mio dubbio è: saprò calcolare bene la concentrazione degli oligo liofilizzati per poi diluirli in maniera corretta per una pcr? 
Si certo!!!  Niente di difficile come vedi! Niente di difficile come vedi!
(l'unica cosa non puoi parlare di "concentrazione" di oligo liofilizzati, è una quantità! La concentrazione è quella che otterrai dopo averli risospesi!)
  |
 |
|
|
stef2
Nuovo Arrivato

21 Messaggi |
 Inserito il - 16 maggio 2008 : 08:41:27 Inserito il - 16 maggio 2008 : 08:41:27




|
Ciao!!!
Ti ringrazio tanto e sei stata esaustiva come sempre!!!!
a presto[:ciao  ] ] |
stef2 |
 |
|
| |
Discussione |
|









