| Autore |
Discussione |
|
|
BioGiac
Nuovo Arrivato

10 Messaggi |
 Inserito il - 14 gennaio 2019 : 23:40:39 Inserito il - 14 gennaio 2019 : 23:40:39



|
Buonasera a tutti
Sto studiando un paper su cui poi dovrò preparare una presentazione (nello specifico questo articolo:
https://www.ncbi.nlm.nih.gov/pubmed/26940867/
Sto trovando però alcune difficoltà nel capire le heat map che vi pongo qui sotto.
Immagine:
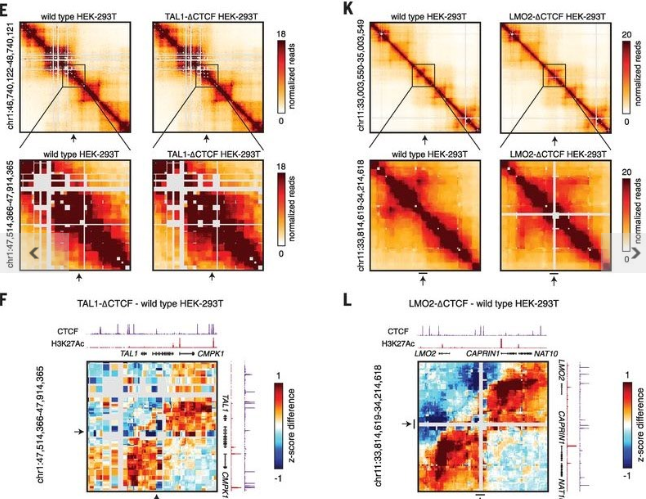
609,93 KB
Fondamentalmente il paper parla del ruolo degli "insulated neighborhoods" (IN) nella repressione trascrizionale di proto-oncogeni coinvolti nella T cell Acute Lymphoblastic Leukemia (T-ALL). Loro quindi introducono tramite CRISPR/Cas9 una delezione nel sito di legame dei CTC Factors (CTCF)che porta alla distruzione dell'insulated neighborhood e quindi porta all'attivazione dell'oncogene (nell'immagine sono riportati TAL1 e LM02).
Tramite queste heatmap (ottenute tramite tecnica 5C) loro supportano l'ipotesi per cui la rimozione del sito di legame per le CTCF nel confine dell'IN che contiene questo gene, porta alla distruzione dell'IN in quanto successivamente alla delezione aumentano le interazioni tra le porzioni interne e quelle esterne a questo loop.
Questo è quanto ho capito ma vorrei imparare a leggere queste heatmap.
Qualcuno potrebbe illuminarmi?
PLS HELP
Grazie in anticipo comunque a chi mi risponderà.
|
|
|
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 15 gennaio 2019 : 09:38:43 Inserito il - 15 gennaio 2019 : 09:38:43




|
Capisco il tuo sconforto, ad una prima occhiata non ci si capisce mai nulla ahah.
In generale queste heatmap le leggi nel seguente modo: i due assi (lati del quadrato) rappresentano una regione cromosomica che viene indicata a lato di ogni heatmap. Quindi hai tutte le posizioni (in basi o piccoli segmenti di DNA)lungo quella regione su entrambi gli assi. Al posto del "terzo asse" hai invece appunto le gradazioni di colore proprie di una heatmap. Nel caso di questi esperimenti si tratta del numero di reads che cadono in quella "coordinata", un po' come a battaglia navale. Mi spiego meglio: la diagonale del quadrato sarà necessariamente una riga continua di reads, perché rappresenta ogni segmento cromosomico contro se stesso, dove avrai tantissime reads che mapperanno con se stesse perché derivanti dalla stessa regione, quindi con il massimo "score" (colore più scuro). Ma la diagonale in sè è la parte meno interessante dell'esperimento (la più ovvia). Il grafico lo leggi partendo da un qualsiasi punto di un qualsiasi lato del quadrato, quindi scorri all'interno della heatmap; poniamo che parti da un punto a caso del lato superiore del quadrato, entri nella heatmap e scorri in basso verticalmente. A ogni posizione cromosomica che incontri lungo questa "discesa" (corrispondente alle posizioni sul lato destro del quadrato) la heatmap ti associa una gradazione di colore che ti rappresenta il "livello" della coassociazione fisica delle due posizioni corrispondenti a quel punto della heatmap stessa. Quindi se vedi degli spot o triangoli scuri fuori dalla diagonale questi corrispondono a regioni cromosomiche che si trovano co-associate fisicamente in cellula.
E' un po più chiaro? |

|
 |
|
|
BioGiac
Nuovo Arrivato

10 Messaggi |
 Inserito il - 15 gennaio 2019 : 17:21:16 Inserito il - 15 gennaio 2019 : 17:21:16



|
In primis grazie mille dell'aiuto.
Quindi coassociate fisicamente nel senso che si trovavano più o meno nello stesso punto?
Cioè quindi nel momento in cui visualizzo questa mappa: nei punti più scuri (che rappresentano quindi punti di intersezione tra varie porzioni interne alla porzione cromosomica scelta e indicata al lato sinistro?) posso dire che le due regioni che si incrociano (stile battaglia navale) il livello di coassociazione (quindi potremmo dire il livello di interazione tra le due porzioni?) è maggiore? e di maggiore interesse quindi sono le porzioni che si discostano di più da questa diagonale giusto?
Spero di non aver fatto troppa confusione ahah
Per quanto riguarda invece le heatmap con lo z-score (cosa sarebbe?) invece come si leggono?
Grazie ancora dell'aiuto  |
 |
|
|
Geeko
Utente
 

Città: Milano

1043 Messaggi |
 Inserito il - 16 gennaio 2019 : 08:49:46 Inserito il - 16 gennaio 2019 : 08:49:46




|
Si è proprio come hai riassunto tu. Per capire meglio il significato dell'esperimento devi vedere bene la tecnica in sè. Il principio è che se nel nucleo due segmenti di DNA si trovano in stretto contatto fisico (si "toccano" magari non direttamente, ma indirettamente grazie a complessi proteici) questi vadano a formare un unico prodotto di ligazione pur essendo lontani a livello di sequenza primaria (1D vs 3D). Quindi ottieni reads da entrambe le regioni coinvolte e provenienti da un unico prodotto di ligazione.
Per gli z-score non so dirti molto, ma sicuramente è un adattamento matematico per rendere più evidenti delle differenze tra due campioni. E' un po' come riassumere due grafici indipendenti in uno solo. Infatti come vedi si parla di "z score difference", quindi è come se avessero "sottratto" i segnali tra due heatmaps. In soldoni più una regione è rossa e più è indice di differenza di contatti intracromosomici tra i due campioni messi a confronto (nel caso specifico cellule delete dell'insulator e cellule wild-type. Si evidenzia infatti che il grosso delle differenze dopo il CRISPR dell'insulator cadono proprio in corrispondenza del locus interessato (TAL1 e LMO2). |

|
 |
|
| |
Discussione |
|







