| Autore |
Discussione |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 01 febbraio 2014 : 17:20:34 Inserito il - 01 febbraio 2014 : 17:20:34




|
Salve a tutti,
non sono un Biologo ma un esperto di grafica 3D (con laurea in questo settore). Sono da sempre interessato alla medicina di laboratorio e alla biologia e sto riqualificandomi in questo ambito.
Vorrei usare la mia esperienza di grafica 3D e Rendering per lavorare (anche per piacere personale) sulla modellazione di proteine.
Volevo da voi qualche consiglio su quali software usare e se ci sono dei corsi che si possono seguire sul Molecular Modelling (anche in inglese ovviamente).
Attualmente utilizzo senza troppi problemi i seguenti software:
BioBlender
VMD
SWISSPDBViewver
PyMOL
3D Studio Max (Rendering e Video)
Non ho problemi con la visualizzazione delle proteine in 3D e con il relativo Rendering. Ho piuttosto problemi a simulare il comportamento delle molecole proteiche inserite in un fluido.
So che dovrei usare software appositi tipo GROMACS ma non sono riuscito neanche ad installarlo.
Il mio scopo (scusate la banalità della domanda) è quello di inserire la proteine in un fluido ad una certa temperatura e pressione e realizzare un video (renderizzato attraverso software professionali di rendering quali 3DStudio Max o Maya) in cui si vede la reazione della proteina. Ancora più interessante è per me vedere l'interazione tra due proteine (esempio vedere la trasformazione fisica e morfologica della pompa sodio/potassio che cambia conformazione quando cede sodio e acquista potassio).
Qualcuno può chiaririmi questi "dubbi" e darmi consigli su come poter procedere per raggiungere lo scopo?
Grazie
Saluti
Luca
|
|
|
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
 Inserito il - 02 febbraio 2014 : 15:31:01 Inserito il - 02 febbraio 2014 : 15:31:01





|
ehm, credo ci sia un pochino di confusione...
Citazione:
Attualmente utilizzo senza troppi problemi i seguenti software:
BioBlender
VMD
SWISSPDBViewver
PyMOL
3D Studio Max (Rendering e Video)
Questo knowledgebase va bene per visualizzare molecole, non per fare modelling nell'accezione della chimica computazionale...
Citazione:
So che dovrei usare software appositi tipo GROMACS ma non sono riuscito neanche ad installarlo.
...e purtroppo e' questo quello che si usa in questo campo, o meglio ci sono anche altre alternative come NAMD o AMBER (ma quest'ultimo e' a pagamento). Se possibile potrei darti una mano nell'installazione di GROMACS.
Citazione:
Il mio scopo (scusate la banalità della domanda) è quello di inserire la proteine in un fluido ad una certa temperatura e pressione e realizzare un video (renderizzato attraverso software professionali di rendering quali 3DStudio Max o Maya) in cui si vede la reazione della proteina.
...mmh, se questo e' il caso ti potrei girare qualcuna delle traiettorie di simulazioni che ho fatto, se dici di saper usare VMD sicuramente le carica.
Citazione:
Ancora più interessante è per me vedere l'interazione tra due proteine (esempio vedere la trasformazione fisica e morfologica della pompa sodio/potassio che cambia conformazione quando cede sodio e acquista potassio).
Spiacente deluderti ma questo genere di cose e' impossibile vederle, in primis per il fatto delle scale dei tempi. Le simulazioni tipicamente campionano tempi sui nanosecondi, attualmente con le piattaforme di calcolo piu' potenti si arriva ai millisecondi (e questo solitamente e' l'ordine dei tempi su cui indagare i cambiamenti morfologici di cui parli). I filmati che potresti aver visto in giro sono di norma ricostruzioni piu' o meno attendibili sulla base di indizi e tecniche un po' piu' astratte/indirette, non rappresentano assolutamente il fenomeno meccanicistico strettamente parlando, in modo neanche lontanamente fedele. |
http://www.linkedin.com/in/dariocorrada |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 15:41:10 Inserito il - 02 febbraio 2014 : 15:41:10




|
Ciao Korda,
ti ringrazio innanzitutto per le tue preziose informazioni.
Per me andrebbe bene anche soltanto inserire la proteina in un fluido per vederne la reazione.
Esempio: se io volessi vedere la pompa Na+/K+ "all'opera" cioè mentre accetta Na+ dall'ambiente extracellulare per rilasciare K+ fuori dalla cellula e quindi vedere il suo cambiamento morfologico (quando cioè la proteina cambia conformazione "fisica" per rilasciare ed accettare ioni) come dovrei fare? Intendo dire una rappresentazione non scientificamente impeccabile ma neanche troppo improvvisata. Cioè mi piacerebbe realizzare un video in cui si vede la proteina che cambia conformazione assumendo una conformazione scientificamente corretta (anche se non avviene nei tempi scientificamente esatti e anche se, ad esempio, gli ioni vengono rappresentati come piccole palline senza un effettivo peso e dimensione).
Quello che io immaginavo era avere una serie di file .pdb in diverse conformazioni fisiche e poi INTERPOLARE i movimenti generando così un'animazione visivamente efficace. Ma come posso ottenere i diversi .pdb della proteina in movimento? Vorrei che il movimento fosse "reale" cioè non qualcosa di improvvisato!
Se tu mi potessi dare una mano con l'installazione di GROMACS te ne sarei veramente grato perchè avrei trovato un paio di "tutorials" che mi aiuterebbero a fare qualcosina (di molto semplice) su gromacs ma non riuscendo ad installarlo non so proprio come procedere!!!
Grazie ancora! |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
 Inserito il - 02 febbraio 2014 : 15:45:37 Inserito il - 02 febbraio 2014 : 15:45:37





|
Citazione:
attualmente con le piattaforme di calcolo piu' potenti si arriva ai millisecondi
mi correggo, microsecondi... |
http://www.linkedin.com/in/dariocorrada |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 15:55:59 Inserito il - 02 febbraio 2014 : 15:55:59




|
Grazie per la precisazione!
Hai qualche modo chiaro che mi aiuti ad installare gromacs?
Ho provato per 3 giorni senza riuscirci. Non sono abituato a installare programmi da righe di comando. Sono purtroppo abituato a farlo tramite file .exe quindi eseguibili automaticamente.
In alternativa sarei disposto anche a retribuire per avere una mano all'installazione di Gromacs. |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
 Inserito il - 02 febbraio 2014 : 15:57:28 Inserito il - 02 febbraio 2014 : 15:57:28





|
Citazione:
Per me andrebbe bene anche soltanto inserire la proteina in un fluido per vederne la reazione.
Per questo potrei girarti qualche mia traiettoria che tu puoi vedere con VMD (cosi aggireresti anche lo sbattimento di installare GROMACS).
Citazione:
Esempio: se io volessi vedere la pompa Na+/K+ "all'opera" cioè mentre accetta Na+ dall'ambiente extracellulare per rilasciare K+ fuori dalla cellula e quindi vedere il suo cambiamento morfologico (quando cioè la proteina cambia conformazione "fisica" per rilasciare ed accettare ioni) come dovrei fare?
Non mi sono mai occupato di questo genere di sistemi, dovresti fare un po' di ricerca bibliografica (su PubMed ad esempio) ed eventualmente contattare gli autori per chiedergli se possono rilasciarti un po' di materiale.
Citazione:
Quello che io immaginavo era avere una serie di file .pdb in diverse conformazioni fisiche e poi INTERPOLARE i movimenti generando così un'animazione visivamente efficace. Ma come posso ottenere i diversi .pdb della proteina in movimento?
Una volta avute le traiettorie credo si possa fare in VMD con qualche scriptino.
Citazione:
Se tu mi potessi dare una mano con l'installazione di GROMACS te ne sarei veramente grato perchè avrei trovato un paio di "tutorials" che mi aiuterebbero a fare qualcosina (di molto semplice) su gromacs ma non riuscendo ad installarlo non so proprio come procedere!!!
Ti aiuterei volentieri se mi dicessi su quale passo dell'installazione non riesci a procedere. Pero' ti avverto fin da subito che, anche con la workstation piu' potente sul mercato, produrre le simulazioni che ti interessano richiederebbero tempi di calcolo biblici (per questo si usano cluster e supercomputer). |
http://www.linkedin.com/in/dariocorrada |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
 Inserito il - 02 febbraio 2014 : 15:59:34 Inserito il - 02 febbraio 2014 : 15:59:34





|
Citazione:
Sono purtroppo abituato a farlo tramite file .exe quindi eseguibili automaticamente.
In alternativa sarei disposto anche a retribuire per avere una mano all'installazione di Gromacs.
Fermi tutti, stiamo parlando di installare GROMACS in un ambiente Windows? |
http://www.linkedin.com/in/dariocorrada |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 16:12:28 Inserito il - 02 febbraio 2014 : 16:12:28




|
| Si, esattamente purtroppo!:( |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 16:17:43 Inserito il - 02 febbraio 2014 : 16:17:43




|
Citazione:
Messaggio inserito da kORdA
Citazione:
Per me andrebbe bene anche soltanto inserire la proteina in un fluido per vederne la reazione.
Per questo potrei girarti qualche mia traiettoria che tu puoi vedere con VMD (cosi aggireresti anche lo sbattimento di installare GROMACS).
Ti ringrazio, sarebbe molto utile per me, magari! Il mio fine sarebbe comunque quello di realizzare autonomamente qualche semplice traiettoria. Ad esempio ne ho vista una in cui si vedeva la molecola di glucosio attraversare una proteina di membrana trasportatore di glucosio. Ecco, questo genere di cose sarebbero interessanti da realizzare. Quella di cui parlo è esattamente questa: http://vimeo.com/64064016
Citazione:
Esempio: se io volessi vedere la pompa Na+/K+ "all'opera" cioè mentre accetta Na+ dall'ambiente extracellulare per rilasciare K+ fuori dalla cellula e quindi vedere il suo cambiamento morfologico (quando cioè la proteina cambia conformazione "fisica" per rilasciare ed accettare ioni) come dovrei fare?
Non mi sono mai occupato di questo genere di sistemi, dovresti fare un po' di ricerca bibliografica (su PubMed ad esempio) ed eventualmente contattare gli autori per chiedergli se possono rilasciarti un po' di materiale.
A questo non ci avevo pensato ma credo che non essendo io ne medico, ne biologo ne chimico nessuno mi rilasci materiale! :(
Citazione:
Quello che io immaginavo era avere una serie di file .pdb in diverse conformazioni fisiche e poi INTERPOLARE i movimenti generando così un'animazione visivamente efficace. Ma come posso ottenere i diversi .pdb della proteina in movimento?[/quote]
Una volta avute le traiettorie credo si possa fare in VMD con qualche scriptino.[/quote]
Per l'interpolazione non ho problemi perchè da VMD converto i vari files .pdb in files .obj da importare all'interno di 3D Studio Max (software potente di Rendering) e da li provvedo all'interpolazione.
|
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 16:21:02 Inserito il - 02 febbraio 2014 : 16:21:02




|
| Quella pagina l'ho riletta almeno 100 volte. Non è dura, è praticamente impossibile! :( Quindi chi ha windows non può usare gromacs? :( |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
 Inserito il - 02 febbraio 2014 : 16:30:33 Inserito il - 02 febbraio 2014 : 16:30:33





|
Citazione:
Ti ringrazio, sarebbe molto utile per me, magari! Il mio fine sarebbe comunque quello di realizzare autonomamente qualche semplice traiettoria. Ad esempio ne ho vista una in cui si vedeva la molecola di glucosio attraversare una proteina di membrana trasportatore di glucosio. Ecco, questo genere di cose sarebbero interessanti da realizzare. Quella di cui parlo è esattamente questa: http://vimeo.com/64064016
mah, per quello contatta direttamente Tiziano
Per le traiettorie magari ci si sente in settimana, quando sono in lab e ho il materiale sottomano.
Per installare GROMACS, ti consiglio caldamente di preparare prima una macchina con su una distro Linux installata in modo nativo (magari il tuo PC, ripartizionandolo). Comincia con una Ubuntu, se non hai mai usato un sistema Linux (oltretutto nei repository sono gia' presenti pacchetti .deb, quindi sbattimenti zero per l'installazione) |
http://www.linkedin.com/in/dariocorrada |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 16:44:32 Inserito il - 02 febbraio 2014 : 16:44:32




|
Mai usato Linux ma neanche Ubuntu! Quindi i files .deb sarebbero i corrispondenti .exe di windows? Cioè gli eseguibili che procedono autonomamente all'installazione, corretto?
Grazie comunque. |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 02 febbraio 2014 : 19:37:56 Inserito il - 02 febbraio 2014 : 19:37:56




|
Citazione:
Mai usato Linux ma neanche Ubuntu!
Ubuntu è una "versione" (il termine corretto è distribuzione) di Linux.
I sistemi Linux sono basati sul Linux kernel (=il cuore) del sistema operativo. Esistono diverse versioni, chiamate distribuzioni, adattate a diverse situazioni. Ubuntu è una delle versioni più adatte per chi voglia fare una transizione Windows->Linux (e forse per questo i "puristi" non la amano troppo, ma sono stupide guerre, il bello di Linux è che sei libero di scegliere quello che piace a te!).
Citazione:
Quindi i files .deb sarebbero i corrispondenti .exe di windows?
Non esattamente, ma il concetto è quello.
L'installazione dei programmi sotto Linux è un po' diversa da Windows. In pratica hai i cosiddetti repository, che contengono un'enorme quantità di programmi e ai quali accedi direttamente dal sistema operativo tramite un programma chiamato gestore di pacchetti. Quindi non devi cercare il sito internet, trovare quale versione del programma installare etc., clicchi sul nome del programma e lui ti installa tutto il necessario. Anche gli update dei programmi sono fatti grazie a questo sistema. Ci sono vari formati di pacchetti contenenti i programmi, come .deb, .rpm e altri.
Insomma, l'idea è più o meno la stessa di Google Play per i sistemi Android o Apple Store per i sistemi Apple, anche se lo spirito alla base è molto diverso.
Per il resto ti lascio nelle esperte mani di Dario... :)
PS: se cerchi ispirazione, trovi degli ottimi rendering di proteine su RCSB. Adoro il loro shader cartoon  |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 02 febbraio 2014 : 23:22:05 Inserito il - 02 febbraio 2014 : 23:22:05




|
grazie chick80, mi stai aprendo un mondo! :) Deve essere interessante UBUNTU. Adesso sto provando ad installarlo sul mio computer però senza partizionare che è un po complesso e ho poco tempo. Vediamo che succede!
Belli gli shader cartoon io li realizzo con i software di rendering (tipo Vray, 3dSMax) ecc!
Una cosa non capisco: se uno volesse attribuire un colore a una proteina (tipo, non so, l'emoglobina...) su cosa dovrebbe basarsi per definire una colorazione? Credo che sia una domanda davvero stupida ma non essendo un biologo mi pongo anche le domande stupide! :( |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 03 febbraio 2014 : 08:15:49 Inserito il - 03 febbraio 2014 : 08:15:49




|
Citazione:
Una cosa non capisco: se uno volesse attribuire un colore a una proteina (tipo, non so, l'emoglobina...) su cosa dovrebbe basarsi per definire una colorazione? Credo che sia una domanda davvero stupida ma non essendo un biologo mi pongo anche le domande stupide! :(
E invece no, non è una domanda stupida!
Ovviamente puoi colorare la proteina come piace a te semplicemente per estetica... ma solitamente si usano i colori per dare delle informazioni biologiche.
Ad esempio, l'emoglobina è formata da 4 catene, 2 chiamate alfa e 2 chiamate beta. Potrai quindi colorare le alfa di un colore e le beta di un altro, come ad esempio:
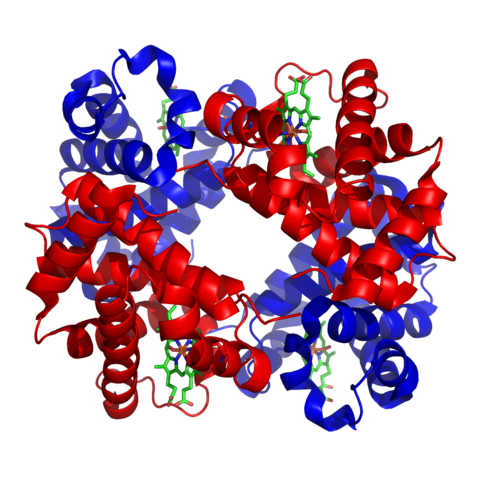
Oppure puoi colorarla a seconda del potenziale elettrostatico, ad es. in questa immagine il blu indica un potenziale negativo, il bianco è neutro ed il rosso è positivo
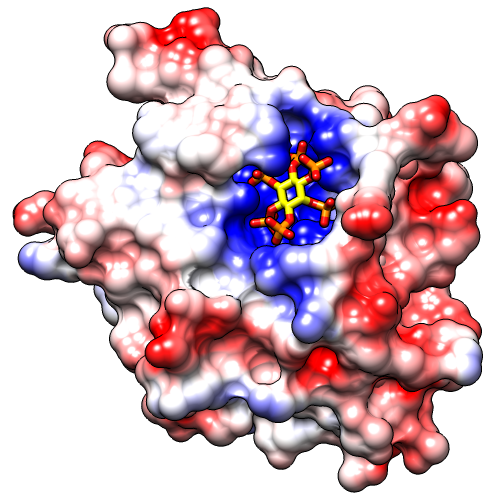
Stesso per l'idrofobicità della molecola

Ci sono sicuramente molti altri parametri che uno può utilizzare per colorare la proteina, ma credo di aver reso l'idea! |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
kORdA
Utente Attivo
  

Prov.: Milano
Città: Monza

1303 Messaggi |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 03 febbraio 2014 : 22:21:37 Inserito il - 03 febbraio 2014 : 22:21:37




|
Citazione:
Messaggio inserito da kORdA
lucaam86, ho appena campionato una traiettoria. Ti ho mandato un PM a riguardo...
Grazie KoRdA, ti ho appena risposto al mess. privato! :) |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 03 febbraio 2014 : 22:25:19 Inserito il - 03 febbraio 2014 : 22:25:19




|
Grazie mille delle info e della disponibilità chick80!
Ho visto che per l'elettro positività / negatività è abbastanza "facile" attribuire una colorazione. Rimane invece un po più complicato per l'idrofobicità. Conosco un programma free chiamato Bioblender che permette di "colorare" la superficie della molecola in base al grado di idrofobicità ma non ho capito poi come posso esportare quel dato in altri software per lavorarlo graficamente meglio.
Comunque molto chiari i tuoi esempi! :) |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 03 febbraio 2014 : 22:38:43 Inserito il - 03 febbraio 2014 : 22:38:43




|
Comunque sono finalmente riuscito a installare GROMACS su Ubuntu (e effettivamente non ci vuole uno scienziato per farlo con Ubuntu!).
Ora seguriò qualche tutorial per prendere confidenza con le varie applicazioni del programma. Anzi, se avete tutorial interessanti da seguire sono ben lieto di conoscerne!
Grazie per il consiglio su Ubuntu! |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 04 febbraio 2014 : 07:49:48 Inserito il - 04 febbraio 2014 : 07:49:48




|
Sempre un piacere convertire qualcuno all'uso del Pinguino :)
E anche Blender è un'ottima scelta! Non conoscevo Bioblender, interessante! Tra l'altro non so se utilizza il motore interno di Blender per il rendering, ma credo che accoppiandolo con LuxRender possa dare dei risultati molto molto interessanti. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
lucaam86
Nuovo Arrivato

94 Messaggi |
 Inserito il - 04 febbraio 2014 : 10:45:18 Inserito il - 04 febbraio 2014 : 10:45:18




|
Citazione:
Messaggio inserito da chick80
Sempre un piacere convertire qualcuno all'uso del Pinguino :)
E anche Blender è un'ottima scelta! Non conoscevo Bioblender, interessante! Tra l'altro non so se utilizza il motore interno di Blender per il rendering, ma credo che accoppiandolo con LuxRender possa dare dei risultati molto molto interessanti.
Si, credo che Bioblender utilizzi il motore interno di Blender per renderizzare le immagini. Tuttavia io ho sempre utilizzato VRAY sotto 3D Studio Max, è praticamente un motore di rendering molto versatile (un po complicato da impostare ma se ci prendi la mano va molto bene). Con 3DS Max o Maya si possono fare animazioni biologiche davvero di impatto.. |
 |
|
| |
Discussione |
|







