Quanto è utile/interessante questa discussione:
| Autore |
Discussione |
|
|
valderica
Nuovo Arrivato

Prov.: Avellino

36 Messaggi |
 Inserito il - 10 marzo 2010 : 13:51:42 Inserito il - 10 marzo 2010 : 13:51:42




|
ciao a tutti!
sono alle prese con una caratterizzazione bioinformatica di questa regione cromosomica murina chr7:109006845..109019302.
sono andata su UCSC.ho scaricato la sequenza di DNA in formato FASTA che ho copiato e incollato su http://www.ebi.ac.uk/Tools/emboss/cpgplot/index.html.
fin qua tutto liscio ma...
purtroppo non riesco a caricare l'immagine per farvi vedere quello che ho combinato...
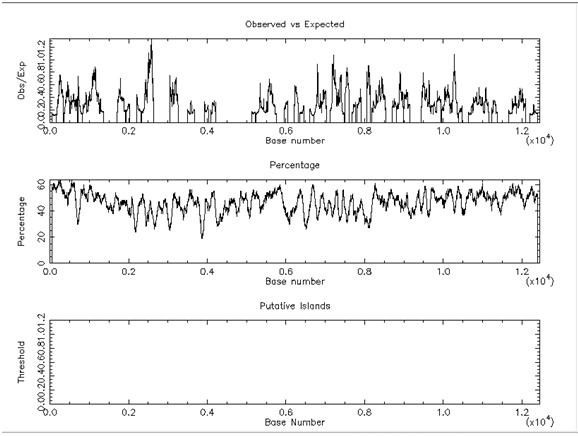
comunque in teoria si ottengono 3 grafici.
nel primo viene visualizzato il rapporto osservati/attesi.il secondo indica la percentuale di GC. infine nel terzo grafico sono riportate le isole CpG rispetto alla soglia e riporta le potenziali isole CpG che superano la soglia. il risultato della mia ricerca è che nel terzo grafico non ottengo nulla,cioè non viene visualizzata neanche una barra,per intenderci,come invece mi sapetterei. il risultato della mia ricerca è che nel terzo grafico non ottengo nulla,cioè non viene visualizzata neanche una barra,per intenderci,come invece mi sapetterei.
sbaglio da qualche parte? è verosimile che non venga visualizzato nulla nel terzo grafico? quindi non ci sono isole CpG in quella regione cromosomica? mi sa che non può essere così mi sa che non può essere così
c'è qualche luminare della bioinformatica che mi aiuta a capirci qualcosa?
grazie
Immagine:
61,66 KB
|
|
|
|
|
valderica
Nuovo Arrivato

Prov.: Avellino

36 Messaggi |
 Inserito il - 10 marzo 2010 : 13:57:30 Inserito il - 10 marzo 2010 : 13:57:30




|
ah,comunque l'immagine incriminata a cui mi riferivo è quella in fondo al mio messaggio precedente. alla fine sono riuscita a caricarla alla fine sono riuscita a caricarla
ho visto alla fine ALLEGA FILE... |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Città: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 10 marzo 2010 : 14:41:06 Inserito il - 10 marzo 2010 : 14:41:06





|
Non é colpa tua, vuol dire che non vi sono isole CpG nella regione osservata.
In effetti se vai su UCSC e provi ad aggiungere la track per RefSeqGenes su questa sequenza vedi che si tratta di una regione codificante, che comprende un gene dall'inizio alla fine, mentre le CpG islands generalmente sono a monte del promotore di un gene, non dentro il trascritto: quindi i tuoi risultati sono verosimili.
Fossi in te, proverei a lanciare il programma prendendo la sequenza a monte del gene, e vedere se vi sono risultati interessanti. |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
valderica
Nuovo Arrivato

Prov.: Avellino

36 Messaggi |
 Inserito il - 10 marzo 2010 : 17:49:29 Inserito il - 10 marzo 2010 : 17:49:29




|
Benone almeno in questo non ho colpa almeno in questo non ho colpa ! !
scherzi a parte, genscan,infatti, predice un solo gene!
incredibile!a volte quando i risultati che si ottengono non combaciano con quelli che uno si aspetta,si è portati subito a pensare di aver sbagliato tutto per un esercizio di bioinformatica come nella vita,o no? per un esercizio di bioinformatica come nella vita,o no? mai una volta che si pensi,invece,che quello che si sta facendo è ok mai una volta che si pensi,invece,che quello che si sta facendo è ok però quando si fanno le esercitazioni sembra che fili tutto,e poi all'esame...ZACk però quando si fanno le esercitazioni sembra che fili tutto,e poi all'esame...ZACk Bhò?! Bhò?!
ah!visto che sei un luminare della bioinformatica ,approfitto. ,approfitto. come si fa a trovare i siti di splicing alternativo?questa è una delle tante cose che continuo a non capire come si fa a trovare i siti di splicing alternativo?questa è una delle tante cose che continuo a non capire diciamo che il prof non riusciva ad essere tanto esauriente come i moderatori di molecular lab diciamo che il prof non riusciva ad essere tanto esauriente come i moderatori di molecular lab
se non si fosse capito mi sto preparando per l'esamone di bioinformatica
comunque,captatio benevolentiae a parte...dallolio_gm mi daresti qualche sincero buon motivo per studiare la bioinformatica? all'uni sono stati in grado di farmene disamorare all'uni sono stati in grado di farmene disamorare
grazie  |
 |
|
| |
Discussione |
|
|
|
Quanto è utile/interessante questa discussione:
| MolecularLab.it |
© 2003-24 MolecularLab.it |
 |
|
|











 però quando si fanno le esercitazioni sembra che fili tutto,e poi all'esame...ZACk
però quando si fanno le esercitazioni sembra che fili tutto,e poi all'esame...ZACk