| Autore |
Discussione |
|
|
qxc
Nuovo Arrivato

9 Messaggi |
 Inserito il - 03 maggio 2010 : 23:06:05 Inserito il - 03 maggio 2010 : 23:06:05




|
Salve a tutti .
Ho disperatamente bisogno di aiuto.
Utilizzando BIOEDIT (software utilizzato in ambito Bioinformatico) dovrei
confrontare la sequenza del mtDNA (limitata a 34bp) delle 2 specie di orsi seguenti :
Ursus arctos e Ursus maritimus.
Ho utilizzato il database di sequenze nucleotidiche consultabile all'indirizzo : http://www.ncbi.nlm.nih.gov/
scaricando le sequenze in formato FASTA.
Con grande sorpresa ho notato che il confronto tra le sequenze di
nucleotidi tra Ursus arctos e Ursus maritimus non evidenzia
similaritą.
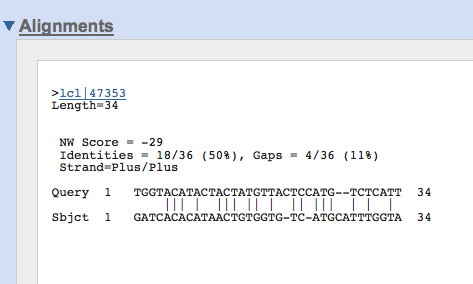
sequenza (34 bp) Ursus arctos
TGGTACATACTACTATGTTACTCCATGTCTCATT
sequenza (34 bp) Ursus maritimus
GATCACACATAACTGTGGTGTCATGCATTTGGTA
Cio' č assurdo in quanto :
Ursus arctos (ORSO BRUNO) č il progenitore di Ursus maritimus (ORSO
POLARE / ORSO BIANCO)
Cioč in pratica si dovrebbe rilevare a vista una grande similaritą tra i due , ma a quanto pare le
due sequenze sono molto diverse.
Il dna č quello mitocondriale.
In fase di ricerca ho raffinato la query scegliendo :
LIMITS poi organism poi molecule-->genominc dna/rna poi gene location mitocondrion.
Mi dite per cortesia dove sbaglio? Oppure se č tutto ok? (non credo ...io penso)
Grazie molte
|
|
|
|
|
domi84
Moderatore
  

Cittą: Glasgow

1724 Messaggi |
 Inserito il - 03 maggio 2010 : 23:41:16 Inserito il - 03 maggio 2010 : 23:41:16




|
Ho una serie di cose da dire : :
A) Bioedit č vecchio (informaticamente parlando), del 2005, quindi non so quando convenga usarlo ancora
B) Non l'ho mai usato ma
Citazione:
LIMITS poi organism poi molecule-->genominc dna/rna poi gene location mitocondrion.
non capisco perchč gli dai tutti questi parametri. Deve solo fare un allineamento tra 2 sequenze
C) prova qualcosa tipo: CLC sequence viewer o T-coffee
D) ho provato con T-coffee e ho avuto dei nucleotidi conservati...quindi una similaritą c'č... |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
Vanilla Sky
Utente Junior



204 Messaggi |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Cittą: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 04 maggio 2010 : 09:58:11 Inserito il - 04 maggio 2010 : 09:58:11





|
Citazione:
mtDNA (limitata a 34bp)
nota che la sequenza di tutto il mtDNA di una specie é ben piu' lunga di 34 bp: ne stai allineando solo una parte, che probabilmente corrisponde a un marcatore come l'HVR che viene utilizzato appositamente per distinguere tra specie diverse. |
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
qxc
Nuovo Arrivato

9 Messaggi |
 Inserito il - 04 maggio 2010 : 23:29:27 Inserito il - 04 maggio 2010 : 23:29:27




|
Grazie .
Quindi la mia query attraverso NCBI era corretta e magari doveva solamente prendere in considerazione
un numero di bp maggiore ( 100 forse )
Magggiore č il numero di nucleotidi presi in considerazione e maggiore č la probabilita di trovare
similaritą tra le due specie ...mi sbaglio?
Il marcatore HVR , non ho chiaro il suo significato .
Mi spieghi?
Grazie
(ti ho anche inviato una email ..grazie ancora )
FAUSTO |
 |
|
|
Ricciola
Nuovo Arrivato

38 Messaggi |
 Inserito il - 05 maggio 2010 : 13:34:32 Inserito il - 05 maggio 2010 : 13:34:32




|
ciao!
io penso, come gli altri, che 34bp siano davvero troppo poche per fare un confronto.
Se non sono indiscreta, per caso stai studiando il DNA barcoding?Per esempio in questo la lunghezza del marcatore molecolare del mtDNA consigliata č circa 650bp.
Tieni presenti che i mtDNA per vari motivi sono soggetti a maggior pressione evolutiva e non hanno efficenti sistemi di riparazione, contrariamente ai nuDNA, pertanto puoi trovare enormi differenze nella sequenza nucleotidica ma sequenze amminoacidiche identiche!
Ah, a titolo informativo, io gli allineamenti li faccio con clustalX e poi apro i files con BioEdit.
Facci sapere
Paola |
 |
|
|
qxc
Nuovo Arrivato

9 Messaggi |
 Inserito il - 06 maggio 2010 : 23:21:47 Inserito il - 06 maggio 2010 : 23:21:47




|
Ringrazio per le risposte.
Non dispongo di una conoscenza cosi profonda come la vostra.
Il mio intento č quello di poter dimostrare , attraverso Bioedit, (il prof vuole quel programma) che l'orso polare č di per sč l'evoluzione del bruno (ursus arctos) .
La cosa ancora strana čla seguente.
Prendendo ancora :
sequenza (34 bp) Ursus arctos
TGGTACATACTACTATGTTACTCCATGTCTCATT
e stavolta
sequenza (34 bp) Tremarctos ornatus
GATCACACATAACTGTGGTGTCATGCATTTGGTA
Mi dite perchč tra i due esiste una similaritą uguale a quella che esiste che tra Ursus arctos e Ursus maritimus ?
Da notare che Tremarctos ornatus si trova in America meridionale quindi molto distante da "brown bear".
Non dovrebbe esserci una similaritą minore?
Grazie
|
 |
|
|
domi84
Moderatore
  

Cittą: Glasgow

1724 Messaggi |
 Inserito il - 07 maggio 2010 : 08:49:55 Inserito il - 07 maggio 2010 : 08:49:55




|
La sequenza che hai scritto di Tremarctos ornatus č identica a Ursus maritimus. Penso tu abbia sbagliato a scrivere.
Comunque il fatto che due specie siano geograficamente distanti non implica che siano anche molto evolutivamente distanti....... |
Il mio blog: http://domi84.blogspot.com/
Le foto che ho scattato... |
 |
|
|
dallolio_gm
Moderatore
  

Prov.: Bo!
Cittą: Barcelona/Bologna

2445 Messaggi |
 Inserito il - 07 maggio 2010 : 10:13:30 Inserito il - 07 maggio 2010 : 10:13:30





|
34 bp sono troppo pochi per poter dire qualsiasi cosa sulla similaritį tra due dna mitocondriali/specie, e anche 100 non bastano.
Nel DNA mitocondriale, come in quello nucleare, vi sono regioni piu' conservati e regioni piu' variabili. Per esempio, se prendi le sequenze di due immunoglobuline di due linfociti dello stesso individuo, ti renderai conto che sono estremamente differenti... mentre i geni codificanti per gli istoni o il sistema di riparazione del DNA sono sempre simili in tutti gli individui.
Nel DNA mitocondriale, tra l'altro, esiste una zone chiamata HVR, estremamente variabile, che viene utilizzata proprio per distinguere tra due individui, per esempio nei test di paternitį o per identificare a chi appartiene un campione di sangue (non so se hai sentito parlare di un concetto chiamato haplogruppo).
Se allinei due sequenze in regioni molto variabili, é normale che vi siano parecchie differenze... dovresti cercare di capire a che regione corrispondono questi 100 nucleotidi che hai trovato, e nel caso cercare una sequenza piu' lunga.
|
Il mio blog di bioinformatics (inglese): BioinfoBlog
Sono un po' lento a rispondere, posso tardare anche qualche giorno... ma abbiate fede! :-) |
 |
|
|
Ricciola
Nuovo Arrivato

38 Messaggi |
 Inserito il - 07 maggio 2010 : 16:11:50 Inserito il - 07 maggio 2010 : 16:11:50




|
qxc 34bp sono una miseria!!!!!!!!!!!!!!!!!!!!!!!!!!
non puoi trarre la benché minima informazione, 500bp č il minimo sindacale, se vai nell'IBOL nessuno analizza sequenze pił corte di 500bp. |
 |
|
| |
Discussione |
|