| Autore |
Discussione |
|
|
biotecman
Nuovo Arrivato

40 Messaggi |
 Inserito il - 27 gennaio 2008 : 17:52:17 Inserito il - 27 gennaio 2008 : 17:52:17




|
qualcuno sa spiegarmi perché in seguito ad estrazione fenolica, a pH acido solo l'RNA rimane in fase acquosa mentre il DNA passa nella fase fenolica?
 
|
|
|
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 28 gennaio 2008 : 00:22:17 Inserito il - 28 gennaio 2008 : 00:22:17




|
questa affermazione non è veritiera, entrambi passano in soluzione acquosa, infatti il fenolo è usato come classico metodo per una purificazione del DNA da proteine fatta a mano.
L'unica differenza tra l'estrazione di DNA e RNA si basa sulla grandezza delle molecole, il DNA genomico durante l'estrazione se non spezzato può essere intrappolato nei lipidi di membrana che precipitano e quindi ritrovato all'interfaccia tra il fenolo e la fase acquosa.
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 28 gennaio 2008 : 03:23:42 Inserito il - 28 gennaio 2008 : 03:23:42





|
Citazione:
Messaggio inserito da Neuroscience
questa affermazione non è veritiera, entrambi passano in soluzione acquosa, infatti il fenolo è usato come classico metodo per una purificazione del DNA da proteine fatta a mano.
        
Vorresti dire che Chomczynski e Sacchi hanno detto una cavolata??? Non penso, visto che dopo 20anni questo metodo di estrazione dell'RNA è tuttora uno dei più utilizzati!!!
Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction.
(Chomczynski e Sacchi, 1987)
The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on.
(Chomczynski e Sacchi, 2006)
C'è una grande differenza tra fenolo acido (pH 4-5) che si usa per separare l'RNA e fenolo tamponato (pH>8) che permette di isolare anche il DNA (in questo caso rimangono entrambi nella fase acquosa!)
Comunque (non ci metto la mano sul fuoco!) la motivazione dovrebbe essere che a pH acido il DNA rimane attaccato agli istoni e quindi intrappolato nella fase organica con questi.
E' possibile poi dopo aver isolato la fase acquosa contenente l'RNA isolare il DNA dalla fase organica innalzando il pH (ad es. aggiungendo NaOH, quello che si fa col Trizol o Trireagent!)
 |
 |
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 28 gennaio 2008 : 19:36:22 Inserito il - 28 gennaio 2008 : 19:36:22




|
bhé... GFPina...
per vedere chi ha ragione basta semplicemente prendere delle cellule e provare... ;-)
io l'ho già fatto e lo faccio tutt'ora mentre ti scrivo, poiché dalle stesse aree cerebrali estraggo DNA e RNA con fenolo acido... sul DNA faccio genotipizzazione, mentre sull'RNA faccio la quantizzazione di espressione.
Qui ci metterei anche la casa sul fuoco  ... ...
Non è necessario fare tanta fatica e consumare tanto materiale... basta fare l'estrazione e poi prelevare la fase acquosa... ci metti un po' di sodio acetato 3M pH 5 (1/10 della soluzione) e poi alcool isamilico volume volume oppure 3 volumi di alcool etilico 95-99%... agiti un po' e miracolosamente si vede il DNA genomico ad occhio nudo, se lo agiti un po' si appallottola e precipita... se in totale ci metti più di 2 minuti, compresa la centrifugata, per questa prova ti pago un caffé 
L'articolo che hai citato, guarda caso è la procedura al GTC che non contiene fenolo, ma ce lo aggiungi dopo. In origine facevo anche questa procedura e quindi la conosco molto bene, la soluzione al GTC è estremamente viscosa.
Se leggi un po' quello che dicono nell'articolo troverai che il DNA non è solubile nel GTC, non nominano la solubilità nel fenolo. La ragione è essenzialmente dovuta alla grandezza del frammento di DNA/RNA... il DNA genomico è lungo centimetri, mentre l'mRNA ha una lunghezza sempre modesta e nel GTC solo queste dimensioni possono solubilizzarsi in maniera efficiente con una semplice agitata. Il DNA genomico invece persino in acqua ha difficoltà a solubilizzarsi se le procedure non sono fatte bene.
Basta infatti che siringhi un po' la soluzione con il DNA oppure vortexarlo, entrambe le procedure rompono il DNA, ed a questo punto il DNA diventa solubile anche in GTC. Idem per il Trizol, le membrane prodotte potrebbero non sequestrare i frammenti di DNA.
Lo stesso principio su cui si fonda l'estrazione del DNA dalle miniprep.... non sono le colonnine a filtrare il genoma dai plasmidi... basta vortexare al momento sbagliato e.... 
Io infatti utilizzo il Fenolo saturo in acqua oppure il Trizol, quest'ultimo guarda caso si usa sia per l'estrazione del DNA che dell'RNA.
A supporto di quanto io abbia già descritto, se proprio non vuoi testare e credermi con le evidenze sperimentali... ti dico che la motivazione dell'uso del pH 4 nell'estrazione di RNA è dovuta solamente e semplicemente per il fatto che un eccesso anche molto modesto di ioni OH in soluzione dà la possibilità al gruppo idrossilico in 2' di deprotonarsi anche per un breve istante ed attaccare il gruppo fosfato che lega i due nucleotidi. Dal legame al fosforo pentamerico che si viene a formare si stacca il legame del fosforo con il nucleotide successivo, rompendo di fatto l'RNA. Questo è un evento raro, diciamo anche 1 su 1000 basi... ma un filamento di mRNA è molto spesso più lungo di 1000 basi quindi l'uso di un fenolo alcalino degraderebbe all'istante tutto l'RNA della soluzione.
Per il DNA la situazione è notevolmente diversa poiché non possiede il gruppo idrossilico, per cui si preferisce un pH più fisiologico ed evitare la minima parte di depurinazione del DNA che può avvenire in ambiente acido... anche qui è un fenomeno raro, ma meglio evitarlo comunque.
Ti faccio sapere se il mio esperimento è riuscito o no, ora mi metto a fare la PCR 
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 gennaio 2008 : 00:24:31 Inserito il - 29 gennaio 2008 : 00:24:31





|
Mi spiace ma purtroppo non posso partecipare alla gara per motivi contingenti, ovvero non ho al momento a disposizione un laboratorio nè tanto meno delle cellule su cui provare!
Comunque lasciamo pure stare il tiocinato di guanidino...
ma trovami un protocollo in cui venga detto che si estrae DNA con fenolo acido!!! A questo punto sono curiosa! 
Io ho anche cercato un po', ma ho solo trovato che il fenolo acido serve per separare RNA da DNA in quanto a pH acido il DNA rimane nella fase organica! Per estrarre il DNA si usa fenolo tamponato!
Tra le varie cose che ho trovato ti segnalo questa che mi sembra esplicativa (non per fare pubblicità alla ditta!):
Application of Prepared Liquid Phenols
dove viene detto:
Citazione:
Determining the pH of Phenol
Depending on the pH of the phenol, DNA will partition into either the organic phase or the aqueous phase (5,6). Thus, it is necessary to accurately determine the pH of phenol solutions.
e c'è anche una bella tabella sul tipo di fenolo e applicazioni.
Gli articoli che citano sono un po' vecchi (e non so se recuperabili), ma questo forse perché è noto da decenni che a pH acido il DNA va nella fase organica... 
Comunque un articolo liberamente accessibile è questo:
A New Method for the Isolation of Deoxyribonucleic Acids: Evidence on the Nature of Bonds between Deoxyribonucleic Acids and Protein
Biochem J. 1957 Jul;66(3):495-504.
Una cosa però non mi è chiara  ... ...
tu usi fenolo acido o fenolo saturo? a che pH?
Citazione:
Messaggio inserito da Neuroscience
Io infatti utilizzo il Fenolo saturo in acqua oppure il Trizol, quest'ultimo guarda caso si usa sia per l'estrazione del DNA che dell'RNA.
P.S. che il Trizol si usa per l'estrazione sia di RNA che di DNA l'avevo detto anch'io (se hai letto?), infatti l'RNA va nella fase acquosa e il DNA in quella organica, dopo aver tolto la fase acquosa si estrae il DNA dall'interfase e dalla fase organica!
 
Aggiungo...
ho trovato anche questo:
Phenol |
 |
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 29 gennaio 2008 : 03:49:53 Inserito il - 29 gennaio 2008 : 03:49:53




|
Mi spiace che tu interpreti questa cosa come una gara,
io la definirei più una curiosità...
Questa procedura è talmente banale che la fanno persino i tirocinanti alle prime armi.
però a tratti sembra che tu te ne intenda ed a tratti invece sembra che tu abbia solo letto qua e là ciò che dici oppure mi stai prendendo in giro
il concetto di fenolo saturo e fenolo acido non sono incompatibili,
infatti il fenolo è acido di per sè e quindi non c'è motivo di chiedere se uso il fenolo acido, sarebbe chiedere se uso l'acqua bagnata.
Ovviamente quando si dice fenolo acido ci si intende che il fenolo sia saturato in acqua acida poiché
il fenolo da solo disidraterebbe la soluzione acquosa che contiene il DNA/RNA.
il pH 4 o pH 8 è definito in base al tampone acquoso con cui è saturato il fenolo.
Quando si dice "ho usato il fenolo acido o alcalino" ci si intende per il fenolo saturato con TrisHCl a pH 4 o pH 8.
Ho letto bene che hai usato la definizione di Trizol, ma hai anche aggiunto
"E' possibile poi dopo aver isolato la fase acquosa contenente l'RNA isolare il DNA dalla fase organica innalzando il pH (ad es. aggiungendo NaOH, quello che si fa col Trizol o Trireagent!)"
cosa che evidentemente non è veritiera, il DNA si trova già nella fase acquosa, variare il pH potrebbe solo a saponificare le membrane liberando del DNA/RNA in esso intrappolato, ma ripeto che non ho mai sentito questa procedura e la ritengo del tutto inutile per l'estrazione del DNA con il Trizol.
E' esperienza quotidiana per chi fa estrazione con il TRizol che il DNA genomico si trovi in soluzione acquosa, difatti per una buona RT-PCR si DEVE necessariamente fare una DNasi ed un controllo negativo per verificare la contaminazione genomica.
E' esperienza quotidiana anche l'estrazione di DNA genomica con il Trizol senza mettere NaOH nell'estratto
Se non puoi farlo tu, sicuramente qualsiasi persona che legge questa discussione e che abbia delle cellule con un po' di trizol può sicuramente testare quello che dico in maniera diretta
Protocollo
Screpare una piastra oppure un tessuto direttamente con TRizol (1 ml per piastra)
Congelare a -80°C e poi riscongelare
Siringare 3 volte delicatamente fino a che la soluzione sia omogenea
aggiungere il cloroformio e alcool isamilico in rapporto 49:1, poi agitare e centrifugare.
La quantità classica di cloroformio è circa 1/5 rispetto al fenolo, ma spesso è necessario arrivare ad 1:1 dipende se si estrae da tessuto o da cellule
Agitare e centrifugare...
Prelevare la fase acquosa facendo bene attenzione a stare lontani dall'interfaccia. Bisogna verificare che nel puntale non ci siano parti di fenolo nel puntale, poi versare in una eppendorf pulita.
Basta aggiungere volume:volume alcool isopropilico, oppure 3 volumi di Alcool etilico 95% lentamente.
Si formerà un anello bianco all'interfaccia alcool:acqua dovuta alla presenza di sali (1° controllo)
poi si inverte lentamente 1 volta e l'anello scompare mentre si vedono delle variazioni di fase (2° controllo).
Invertendo dalla seconda fino alla n volta si vedrà il DNA precipitare per poi arrotolarsi (3° controllo).
A questo punto si centrifuga (o si prende il DNA con un'ansa), si secca un po', si fanno 2 lavaggi con alcool etilico 70%, si tolgono tutte le goccie rimanenti di alcool e poi si solubilizza il DNA in TE pH 7.4, es 50 ul. La soluzione diventerà filamentosa (4° controllo)
Si carica su gel di agarosio 0,8% 1/10 di volume (50 ul) e poi si fa una PCR su genomico per controllo
Sul gel si vedrà una banda molto alta a circa 23.000 basi con una migrazione lentissima (5° controllo).
La PCR ovviamente è la prova del 9  ma bisogna essere certi che non ci siano troppe schifezze nel DNA, es troppo fenolo/cloroformio o proteine. 1 ul di soluzione del DNA dovrebbe bastare. ma bisogna essere certi che non ci siano troppe schifezze nel DNA, es troppo fenolo/cloroformio o proteine. 1 ul di soluzione del DNA dovrebbe bastare.
Credo che ci siano dettagli a sufficienza anche per il più modesto tesista di laboratorio alle prime armi, ma se servono altri dettagli della procedura non abbiate paura a chiedere.
Vi prego di scrivere qua il risultato... bastano 5 minuti per questa prova, più la PCR il cui risultato è scontato se già vedere il DNA sul gel di agarosio.
 |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 29 gennaio 2008 : 05:39:21 Inserito il - 29 gennaio 2008 : 05:39:21




|
Premetto che non uso il Trizol da una vita, comunque quello che mi ricordo io è che il DNA non va in fase acquosa, ma resta nell'interfase/fase organica.
D'altronde il Trizol è fatto per estrarre l'RNA (anche se ci puoi comunque recuperare DNA e proteine), per il DNA Invitrogen produce il DNAzol...
Ad ogni modo invece di litigare la cosa più veloce è vedere cosa dicono i siti delle case produttrici.
Invitrogen:
TRIZOL Reagent is a ready-to-use reagent for the isolation of total RNA from cells and tissues. The reagent, a mono-phasic solution of phenol and guanidine isothiocyanate, is an improvement to the single-step RNA isolation method developed by Chomczynski and Sacchi. During sample homogenization or lysis, TRIZOL Reagent maintains the integrity of the RNA, while disrupting cells and dissolving cell components. Addition of chloroform followed by centrifugation, separates the solution into an aqueous phase and an organic phase. RNA remains exclusively in the aqueous phase. After transfer of the aqueous phase, the RNA is recovered by precipitation with isopropyl alcohol. After removal of the aqueous phase, the DNA and proteins in the sample can be recovered by sequential precipitation.
Allo stesso modo uno può leggere quello che dice il sito della Ambion sul loro TRI Reagent
http://www.ambion.com/techlib/prot/bp_9738.pdf
E qui c'è proprio il disegnino della provetta con l'RNA nella fase acquosa e il DNA/proteine nella interfase/fase organica
Sul sito della Sigma troviamo il "manuale" del Tri-Reagent che dice:
http://www.sigmaaldrich.com/sigma/bulletin/t9424bul.pdf
"The DNA is in the interphase and phenol phase which forms after the addition of chloroform to the
TRI REAGENT in step 2 of sample preparation. After precipitation and multiple washes, the DNA is dissolved in 8 mM NaOH. The solution is neutralized and the DNA is ready for analysis. The resulting DNA is suitable for PCR, restriction enzyme digestion and Southern blotting"
  |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 gennaio 2008 : 13:18:57 Inserito il - 29 gennaio 2008 : 13:18:57





|
Citazione:
Messaggio inserito da Neuroscience
Mi spiace che tu interpreti questa cosa come una gara,
io la definirei più una curiosità...
Come non era una gara??? Ma non c'era in pallio un caffè?
Citazione:
Messaggio inserito da Neuroscience
....per questa prova ti pago un caffé 
No sai più che altro era l'occasione per bermi un buon caffè  , dalle tue parti... qua è tremendo e costa un patrimonio... , dalle tue parti... qua è tremendo e costa un patrimonio...
A parte gli scherzi...
scusa sul fenolo saturo hai ragione!!!
Mi sono accorta adesso di quello che ho scritto... in effetti non aveva alcun senso... perdona la stanchezza di ieri sera!! Ho avuto una giornata abbastanza pesante! Ho avuto una giornata abbastanza pesante!
Comunque volevo semplicemente sapere quale era il fenolo che usi.
Ah giusto per precisare... il fenolo da solo a Tamb. è solido dovresti scaldarlo (che non è una cosa tanto bella!), si usa quindi saturato con acqua o miscelato al cloroformio.
Il fenolo è di per se acido (come dici tu) e può essere portato in soluzione a pH basico.
Ok su questo siamo d'accordo!
L'unico punto che rimane in discussione è cosa succede al DNA in fenolo acido??? 
Allora io purtroppo (come ti dicevo) in questo momento non posso provare il tuo protocollo... quindi non potendo testare sperimentalmente mi rifaccio alla letteratura!
Io ho trovato scritto da qualunque parte che con fenolo acido il DNA va nella fase organica, tu dici che rimane nella fase acquosa... ok potresti aver ragione ma esiste qualche pubblicazione al riguardo?
Altrimenti (non sto scherzando!) ti consiglio di pubblicarlo tu!!!
@chick80: non mi sembra che stessimo litigando... credo che sia una "sana" discussione su due punti di vista diversi che possa servire a molti! Anzi chiunque voglia portare la sua esperienza è il benvenuto!!! Non penso che solo io e Neuroscienze abbiamo fatto etstrazione di RNA!
Comunque con questo rispondo ad entrambi:
la mia affermazione
Citazione:
Messaggio inserito da GFPina
E' possibile poi dopo aver isolato la fase acquosa contenente l'RNA isolare il DNA dalla fase organica innalzando il pH (ad es. aggiungendo NaOH, quello che si fa col Trizol o Trireagent!)
viene appunto dal protocollo de Trizol... non è una mia invenzione:

questo è il link al protocollo completo:
TRIzol
 
----------------------------------------------------------------------------------------------------------------------
Scusate aggiungo una curiosità mia:
ma la differenza tra TRIzol e DNAzol è che nel primo c'è anche il fenolo e nel secondo solo tiocianato guanidinio? (le formulazioni sono protette da patente, ma cosi' parrebbe!)
Comunque sono stati inventati tutti e due da Chomczynski P. ecco le references:
DNAzol: a reagent for the rapid isolation of genomic DNA.
A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. |
 |
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 29 gennaio 2008 : 15:34:16 Inserito il - 29 gennaio 2008 : 15:34:16




|
Caro chick80,
se leggi quello che dicono le case produttrici per kit da miniprep ti diranno di sicuro che con quel kit estrai solo i plasmidi batterici e non il DNA genomico, d'altronde non avrebbe senso fare un kit per estrarre il DNA genomico del batterio... eppure se lo usi "male" il kit delle miniprep estrae anche il DNA genomico del batterio, basta vortexare quando si mette il buffer di lisi oppure il buffer di precipitazione... et voilà esce fuori il DNA genomico del batterio pur usando un kit per miniprep.
Il principio di cui vi sto parlando è lo stesso, la separazione non avviene in quanto uno è DNA e l'altro RNA, entrambi sono solubili più o meno allo stesso modo, ma è la dimensione che fa la differenza, uno è lungo miliardi di basi, l'altro migliaia, è ovvio che con la formazione di membrane precipitate il DNA non possa restare in soluzione, mentre a pH alcalino le membrane sono ionizzate e quindi
Venendo alla discussione in questione, credo che ognuno di noi abbia esplicato abbastanza le proprie idee e le proprie esperienze, andare ancora a puntualizzare farebbe diventare questo thread noioso, petulante e del tutto sterile. Non trovo altra soluzione nel progredire questa discussione filosofica nell'applicarla tecnicamente e verificare quanto è stato detto o affermato dalle rispettive parti.
Chick80, dato che ti sei inserito nella discussione  ed aggiungerei anche in modo imparziale, secondo il mio punto di vista, e dato che un po' di esperienza minima di laboratorio ce l'hai, perché non fai questo esperimento per la gioia del thread e dei posteri ed aggiungerei anche in modo imparziale, secondo il mio punto di vista, e dato che un po' di esperienza minima di laboratorio ce l'hai, perché non fai questo esperimento per la gioia del thread e dei posteri  ? ?
hai bisogno di una piastra o un po' di tessuto, 1 ml di Trizol e 5-10 minuti compresa la migrazione su gel e foto storica da inserire nel thread.
Lo farei io, e la foto ce l'ho, l'ho fatta proprio ieri mentre scrivevo, ma mi risulterebbe difficile essere credibile, credo abbia più impatto se lo facesse qualcuno imparziale o che ci crede poco.
Con questo puntualizzo il fatto che per fare una estrazione di RNA senza DNA ci vuole mooooolta esperienza e moooolta pratica, poiché, fenolo acido o no, se si fa il minimo sbaglio il DNA genomico resta nella fase acquosa e non si ingloba nei lipidi di membrana.
Il punto critico è l'agitazione o la siringazione del tesuto dopo l'aggiunta del cloroformio, se si agita troppo il DNA non rimane intrappolato, se si agita poco non si ha una buona purificazione.
Da quello che dice GFPina invece sarebbe impossibile estrarre DNA con il Trizol senza fare uso di NaOH, a mio avviso l'NaOH serve solo per saponificare i lipidi e liberare il DNA, quando riesci ad intrappolarlo, altri ruoli non ne ha.
Con questo chiudo, poiché altre sarebbero solo parole vane.
P.S.: nonché per la tua simpatia ti pago comunque il caffé ovunque tu sia, dovessi portartelo a domicilio o in trasferta da qualche parte... magari a qualche congresso o altro 
Sarò al prossimo FENS poi al SINS
 |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 gennaio 2008 : 17:32:29 Inserito il - 29 gennaio 2008 : 17:32:29





|
Aggiungo solo che ho recuperato la mia vecchia metodica per isolare l'RNA (anche se io ne avevo usata una versione un po' diversa per adattarla ai microRNA)... ma la cosa divertente è che è praticamente uguale alla tua... però io ottenevo RNA e tu DNA... 
Comunque la posto.
Estrazione RNA
Soluzione Denaturante:
• 4 M isotiocianato di guanidinio (GITC)
• 0.02 M sodio citrato
• 0.5% sarcosyl
• Prima dell’uso aggiungere 0.72#956;l di #946;–ME in 100#956;l si soluzione
1. Omogenizzare il campione nella soluzione denaturante (100#956;l di soluzione per 10mg or 1 × 106 cellule)
2. Aggiungere 1/10 di 2 M sodio acetato
3. Aggiungere 1 volume di fenolo (equilibrato a pH 5.3–5.7 con acido succinico)
4. Aggiungere 1/5 di volume di cloroformio:isoamyl alcohol e vortexare vigorosamente
5. Centrifugare il campione in una microcentrifuga a 13–14000 rpm per 5 minuti.
Dovrebbero essere chiaramente visibili due fasi. Un po’ di DNA e proteine potrebbero essere presenti nell’interfase.
6. Trasferire la fase acquosa superiore contenente l’RNA, facendo attenzione a non toccare l’interfase, in una provetta contenente un volume di isopropanolo. Eliminare la fase inferiore contenente fenolo, proteine e DNA.
7. Centrifugare il campione in una microcentrifuga a 13–14000 rpm per 5 minuti.
8. Rimuovere completamente il surnatante
9. Lavare il pellet in 2 volumi di 75% etanolo
10. Rimuovere completamente il surnatante
11. Lasciar asciugare per 5 minuti
12. Risospendere il pellet in un 1/2 -1/10 volume di H2O RNAsi free
Citazione:
Messaggio inserito da Neuroscience
Da quello che dice GFPina invece sarebbe impossibile estrarre DNA con il Trizol senza fare uso di NaOH,...
Beh non da quello che dico io, ma da quello che c'è scritto da diverse parti...
Comunque chiudo anch'io ma se qualcuno fa qualche esperimento aspettiamo i risultati 
 
P.S. Grazie comunque del caffè anche se non vado a congressi di Neuroscienze e penso che se tu me lo portassi a domicilio si rovinerebbe!  |
 |
|
|
Neuroscience
Utente
 

659 Messaggi |
 Inserito il - 29 gennaio 2008 : 17:53:59 Inserito il - 29 gennaio 2008 : 17:53:59




|
Io dico che estrai sia DNA che RNA come faccio io, l'unica differenza sta quando precipito con alcool prendo con l'ansa il DNA genomico e lo sciolgo a parte, mentre l'RNA continuo la procedura clasica.
Ricordi di aver mai fatto un controllo negativo per la contaminazione di DNA genomico?
oppure hai mai valutato la quantità di DNA genomico che ottenevi? oppure quantizzavi l'RNA retrotrascrivevi e poi direttamente PCR?
Se i tuoi primers potevano amplificare anche un tratto di genoma allora era importante valutare l'eventuale contaminazione. |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 gennaio 2008 : 18:27:50 Inserito il - 29 gennaio 2008 : 18:27:50





|
Il fatto è che tu dici che vedi il gomitolo... io non l'ho mai visto!!!
Che ci possa essere contaminazione da DNA non lo escludo, ma potrebbe anche essere che "parte" del DNA va in soluzione non che sia tutto li, non vedo quindi il controllo come un fattore discriminate.
Controlli negativi ne ho fatti, ma anche trattamento con DNAsi... ho fatto tanti di quelli esperimenti diversi e tante modifiche ai protocolli per adattarli meglio alle mie esigenze che non potrei darti una risposta precisa. Inoltre tutti i miei campioni e i risultati sono rimasti in Italia...
Ma mi riservo di fare una prova appena ne avrò l'occasione! 
Altro al momento non ti posso dire, se non che trovi una marea di materiale in letteratura e su Internet con protocolli (uguali a quello) che dicono che sono specifici per RNA.
ad es questo: Protocol for RNA Isolation From Tissue Samples from: theLabRat (è carino il rattino quanto meno  !) !)
Comunque visto il numero di lettori, anche se nessun altro è intervenuto, devo dire che la discussione ha riscosso successo  |
 |
|
|
Dionysos
Moderatore
  

Città: Heidelberg

1913 Messaggi |
 Inserito il - 29 gennaio 2008 : 23:20:47 Inserito il - 29 gennaio 2008 : 23:20:47





|
Per forza nessun altro è intervenuto, GFPina!!! è stato interpretato come uno scontro fra titani |
Volere libera : questa é la vera dottrina della volontà e della libertà
(F.W. Nietzsche)
Less Jim Morrison, more Sean Morrison!
|
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 29 gennaio 2008 : 23:32:02 Inserito il - 29 gennaio 2008 : 23:32:02





|
Ti ringrazio Dionysos però perché mai???
Penso che ce ne siano di persone competenti qua comunque, non solo studenti!
Io ogni caso se qualcuno (anche alle prime armi) ha avuto esperienze al riguardo o vuole fare la prova è bene accetto io prometto che non lo "mangeremo" se posta!!! 
Al massimo alla fine si va tutti a bersi un caffè! 
(scusate ma quello proprio qua mi manca  ) ) |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 30 gennaio 2008 : 02:31:22 Inserito il - 30 gennaio 2008 : 02:31:22




|
Se leggete bene cosa dice il product sheet:
"RNA remains exclusively in the aqueous phase. After transfer of the aqueous phase, the RNA is recovered by precipitation with isopropyl alcohol. After removal of the aqueous phase, the DNA and proteins in the sample can be recovered by sequential precipitation."
Cioè, loro ti garantiscono che l'RNA rimane solo nella fase acquosa, poi ti suggeriscono di usare le altre fasi per estrarre il DNA. Questo non vuol dire che nella fase acquosa non hai DNA, infatti nello stesso product sheet ti dicono di usare primers a cavallo di introni e di trattare con DNAsi...
Quindi probabilmente è semplicemente un problema di che % resta nel fenolo e che % resta nella fase acquosa.
Comunque io per fare i genotipi facevo così
http://www.molecularlab.it/protocolli/protocol.asp?id=8
|
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
Luca Beretta
Nuovo Arrivato

Città: Milano

12 Messaggi |
 Inserito il - 12 febbraio 2008 : 10:54:07 Inserito il - 12 febbraio 2008 : 10:54:07




|
ragazzi, chicca riguardo al Trizol... 
il trizol non viene prodotto da invitrogen ma da una società (MRC) che ne detiene il patent... lo produce e lo rivende a invitrogen (che lo chiama trizol), ad ambion (che lo chiama tri reagent) etc etc, ma sono tutti lo steeso identico prodotto! Cambia solo il prezzo... Invitrogen la confezione da 100ml la fa pagare da listino 232 euro, Ambion lo stesso prodotto stesso confezionamento 119 euro!

Vi lascio il codice, io lo uso da tempo ed è perfetto: AM9738

NB Ambion ora è di Applied Biosystems |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 12 febbraio 2008 : 13:41:41 Inserito il - 12 febbraio 2008 : 13:41:41





|
Ah ah mi chiedevo come mai fosse stata riaperta questa discussione! 
Luca hai fatto bene a fare questa precisazione, comunque aggiungo che basta cercare TriReagent e saltano fuori una miriade di ditte che lo vendono...
anche Sigma che ne ha addirittura 4 versioni, Genscript...
ma persino la stessa MCR (Molecular Research Center) lo rivende attraverso vari distributori, in Italia BioOptica...
quindi basta cercare per trovare il prezzo migliore! 
Citazione:
Messaggio inserito da Luca Beretta
NB Ambion ora è di Applied Biosystems
     Non me lo ricordare!!! Non me lo ricordare!!!    |
 |
|
|
Topogigio77
Nuovo Arrivato

Città: Milano

37 Messaggi |
 Inserito il - 12 febbraio 2008 : 13:47:17 Inserito il - 12 febbraio 2008 : 13:47:17




|
Grande!  |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 14 marzo 2008 : 23:55:33 Inserito il - 14 marzo 2008 : 23:55:33





|
confermo l estrazione di RNA con Fenolo acido,mentre procedevo mi sono ricordato questo topic...anche perche' si si usa pH basico,gli alcali dovrebbero degradare l RNA
E' uscito una meraviglia |

|
 |
|
|
pinocitosi
Nuovo Arrivato

Prov.: Novara
Città: novara

11 Messaggi |
 Inserito il - 09 novembre 2008 : 22:06:21 Inserito il - 09 novembre 2008 : 22:06:21




|
| Scuate..ma quando mi prendo la fase acquosa con l'RNA e poi lo faccio precipitare con etanolo mi dite come faccio a fare i lavaggi se ho un pellet seminvisbile? |
 |
|
|
chick80
Moderatore
    

Città: Edinburgh

11491 Messaggi |
 Inserito il - 09 novembre 2008 : 22:24:43 Inserito il - 09 novembre 2008 : 22:24:43




|
Si chiama fiducia! 
Il pellet è attaccato al fondo della provetta, se usi un po' di delicatezza vedrai che non si stacca. |
Sei un nuovo arrivato?
Leggi il regolamento del forum e presentati qui
My photo portfolio (now on G+!) |
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 09 novembre 2008 : 22:31:17 Inserito il - 09 novembre 2008 : 22:31:17





|
Ahhhhhh ancora questo post!!! 
Comunque per la tua domanda:
Citazione:
Messaggio inserito da pinocitosi
Scuate..ma quando mi prendo la fase acquosa con l'RNA e poi lo faccio precipitare con etanolo mi dite come faccio a fare i lavaggi se ho un pellet seminvisbile?
Basta che quando centrifughi le provette le metti tutte ordinate in una certa maniera, se le metti con la cerniera rivolta verso l'esterno, così:

(non si vede moltissimo, ma è la foto che ho trovato)
il pellet dopo centrifugazione si troverà sul lato anteriore della provetta in basso:
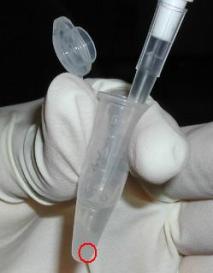
anche se non lo vedi!
In questo modo basta che tu stia attento a non toccare mai col puntale in quel punto! 
  |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 09 novembre 2008 : 23:23:06 Inserito il - 09 novembre 2008 : 23:23:06





|
| io lo vedo sempre e uso la siringa per insulina che e' ancora meglio penso |

|
 |
|
|
GFPina
Moderatore
    

Città: Milano

8408 Messaggi |
 Inserito il - 09 novembre 2008 : 23:37:06 Inserito il - 09 novembre 2008 : 23:37:06





|
Citazione:
Messaggio inserito da Patrizio
io lo vedo sempre ...
Beh se lo vedi sempre si vede che estrai da campioni che ne hanno tanto!  (o hai la vista a raggi X!) (o hai la vista a raggi X!) 
Non sempre è visibile!!! Ma basta usare alcuni accorgimenti per non perderlo anche se è "invisibile"!  |
 |
|
|
Patrizio
Moderatore
  

Città: Barcellona

1915 Messaggi |
 Inserito il - 09 novembre 2008 : 23:46:16 Inserito il - 09 novembre 2008 : 23:46:16





|
no va be ma anche perche' sono bravo 
fin ora ho estratto da Hela e fibroblasti con la multiwell da 6 trasfettati e non |

|
 |
|
|
wiifii
Nuovo Arrivato

3 Messaggi |
 Inserito il - 21 maggio 2009 : 16:11:05 Inserito il - 21 maggio 2009 : 16:11:05




|
| il post è vecchissimo ma solo ora lo consulto se posso azzardare una mia personale ipotesi sulla vicenda(sono una studentella quindi posso anche dire una grande scemenza): forse le cellule prese in esame da neuroscienze erano cellule batteriche il cui dna privo di istoni rimaneva nella fase acquosa con l rna. |
 |
|
|
Discussione |
|













