Durante l´esecitazione verranno utilizzati i browser genomici
Ensembl (www.ensembl.org)
e Genome Browser (http://genome.ucsc.edu)
e verranno esplorate alcune delle loro funzionalitá: In particolare
verranno analizzati i seguenti temi:
- Ricerca di geni mediante parole chiave o mediante similaritá
di sequenza.
- Selezionare l'organismo di interesse
- Per cercare con il nome del gene, inserire nel campo opportuno
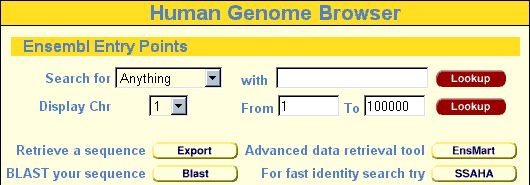
- Per cercare secondo similarità, cliccare su Blast, e
inserire la sequenza in possesso (prova a farlo con la proteina
trovata prima)
- Per sequenze particolarmente corte (20 -30 nucleotidi), SSAHA
permette di avere un algoritmo di ricerca migliore, ottimizzato.
- Estrazione della sequenza di geni omologhi
-
Una volta entrati nella pagina del gene di interesse (COX4-1),
tra le diverse informazioni, c'è anche "Homology Matches"
in cui si trovano gli omologhi al gene.
- Estrazione di informazioni collegate al gene di interesse: (trascritti
alternativi, proteina codificata, famiglie proteiche)
- Questo può essere fatto facilmente dalla scheda per ogni
gene, o direttamente dal Browser:
Scegliete di vedere nella "Genomic
Location" , dalla quale potrete vedere una serie di informazioni
in tre livelli: Overview, Dectail View e Base View. In particolare,
nella Dectail View, si hanno le magiori informazioni riguardo
al gene e a quello che sta attorno. Cliccando sulla barra gialla,
è possibile accedere alle innumerevoli opzioni: visualizzare
le feature di interesse, indicare a quale banca fare riferimento
(o uploadare i propri dati), esportare la regione visualizzata
in diversi formati, passare ad altri GenomeBrowser, e indicare
la dimensione della finestra.
- Estrazione di sequenze genomiche corrispondenti alla regione
a monte del gene di interesse (promotore)
- Nella Genomic Location, provate a muovervi lungo la sequenza,
usando le opzioni di Zoom, o per spostarsi di 1-2Mb, o digitare
manualmente le coordinate.
- Osservate i geni presenti, vicino a COX4 ... passando il mouse
su i diversi geni, è indicato nome e opzioni di visualizazione
relativa al gene su cui si è.
ESECIZI PROPOSTI:
- Su quale cromosoma si trovano i geni per le diverse subunitá
del complesso citocromo ossidasi umano?
- Quale é l´AC RefSeq o EMBL per le sequenze tracritte
(mRNA) di tali subunitá?
- Fai la stessa ricerca nel topo.
- Quali domini sono presenti nella proteina codificata da questo
trascritto?
- Esistono delle forme di splicing alternative?
- Cerca dei membri della stessa famiglia proteica in altri vertebrati
e crea un multiallineamento.
