Tecnica che permette di separare frammenti di acido nucleico, in
funzione del peso molecolare.
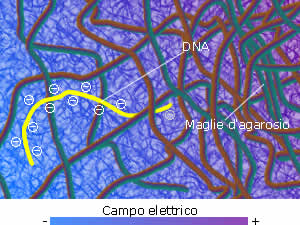
Frammenti di DNA, carichi negativamente per i residui di fosfato,
in un campo elettrico tendono ad andare verso il polo positivo.
Usando una griglia molecolare costituita da agarosio (o acrilammide
se si vuole ottenere una maggiore risoluzione -usata nel sequenziamento-)
lasciato polimerizzare (l'agarosio si scioglie in acqua con temperature
sopra i 70°C, raffreddandosi polimerizza), i frammenti passano
attraverso di essa. Più sono grandi e pesanti e più
fanno fatica restando indietro rispetto a frammenti più piccoli
che passano più velocemnte tra le maglie.
La visualizzazione è garantita da sostanza intercalanti,
come l'etidio di bromuro, che sono fluorescenti se esposte ai raggi
UV. L'uso d un ladder (una serie di frammenti a dimensione nota),
mi permette per confronto di avere informazioni sulle dimensioni
dei frammenti.
L'elettroforesi è una tecnica che si applica sia con DNA
che con RNA, sia a singolo che doppio filamento.
Variando la concentrazione di agarosio ottengo maglie più
o meno fitte, da usare in funzione della dimensione del frammento
(più è piccolo e più migra velocemente) e se
si tratta di un frammento ds o ss.
L'elettroforesi capillare è una elettroforesi che viene eseguita
su una colonna similmente ad una cromatografia.
Tecniche simil-derivate:
SDS-Page
Notehrn Blot
Western Blot
Southern Blot
Ulteriori informazioni sulla Elettroforesi e tecniche derivate
nella sezione Tecniche,
in Laboratorio
