-5° giorno-
Analisi risultati e preparazione minicolture
Poichè la trasformazione delle cellule preparate
dal gruppo è stata svolta con una quantità di DNA
plasmidico non saturante, si può procedere al calcolo dell'efficienza
di trasformazione:
Efficienza trasformazione = ° colonie resistenti all'Amp / ug
DNA plasmidico
Protocollo:
Preparazione all'analisi dei plasmidi ricombinanti:
- Scelta di 2 cloni bianchi (contenti l'inserto, e resistenti
all'Amp)
- Prelievo sterile da clni isolati con ansa sterile, e inoculo
di 5ml di terreno LB+Amp
- Incubazione a 37°C
- Preparazione
di gel d'agarosio per il giorno dopo
Note, risultati e
conclusioni:
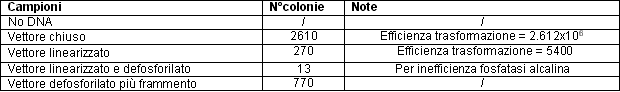
Come aspettato nella piastra in cui non era stato messo DNA non
si è osservata alcuna crescita, essendo il terreno contenente
ampicillina ed il gene per la resistenza all'antibiotico sul plasmide.
Nella piastra con il plasmide chiuso i frammenti alfa, sul plasmide,
e omega, sul menoma batterico, hanno permesso di ottenere una beta
galattosidasi funzionante la quale, reagendo con l'X-gal ha reso
le colonie colorate di blu. La sintesi della beta galattosidasi
è permessa dall'induttore dell'operone LAC IPTG messo nel
terreno. In particolare l'induttore si lega al repressore dell'operone,
rendendo così la produzione di beta galattosidasi costitutivamente
espressa.
Nella piastra contenente il prodotto della ligazione dal vettore
linearizzato si osserva una mediocre crescita. In questo modo possiamo
testare l'enzima ligasi usato.
Poiché la fosfatasi alcalina non è efficiente al 100%
si osserva la presenza di cloni anche nella piastra il cui prodotto
di ligazione era stato il vettore defosforilato e linearizzato.
Nella piastra in cui non vi erano state messe le cellule trasformate
con il prodotto di ligazione tra il vettore defosforilato ed il
frammento si osserva la presenza di colonie bianche, in quanto il
frammento rende la beta galattosidasi inattiva.